| x | x | ||||
 |
|||||
| BACTERIOLOGIE | IMMUNOLOGIE | MYCOLOGY | PARASITOLOGY | VIROLOGIE | |
|
|||||
| EN ANGLAIS | |||||
| IN PORTUGUESE | |||||
| IN SPANISH | |||||
| SHQIP - ALBANIAN | |||||
| IN FARSI | |||||
|
|
|||||
|
Let us know what you think |
|||||
| SEARCH | |||||
|
|
|||||
| Logo image © Jeffrey Nelson, Rush University, Chicago, Illinois and The MicrobeLibrary | |||||
|
Reading: Murray, 6thedition, Chapter 3 |
|||||
|
MOTS CLES |
Identification bactérienne dans un laboratoire de diagnostique versus taxonomie L’isolation et l’identification de bactéries à partir de patients aide au traitement puisque les maladies infectieuses causées par différentes bactéries présentent une variété d’évolution et de conséquences cliniques. L’évaluation de la susceptibilité d’isolats (c'est-à-dire la détermination de la concentration inhibitrice minimale ou MIC) peut aider à la sélection d’antibiotique en vue d’une thérapie. L’isolement de certaines espèces (ou souches) de manière atypique peut suggérer qu’une infection s’est développée à cause de matériel médical contaminé ou de techniques d’asepsie insuffisantes de la part du personnel hospitalier. Lorsqu’on suspecte une infection bactérienne chez un patient, il est habituel d’isoler des colonies visibles de l’organisme en culture pure (sur des boîtes d’agar) puis de préciser l’identification de l’organisme. L’identification est basée sur des principes taxonomiques appliqués à une situation microbiologique clinique. Dans le laboratoire de diagnostique, de nombreux échantillons doivent être traités chaque jour et les résultats obtenus aussi rapidement que possible. Les tests doivent être facilement appris, de faible coût et réalisable rapidement. Ces méthodes classiques pour la spéciation de bactéries sont basées sur des caractéristiques morphologiques et métaboliques. Les tests diagnostiques ont été sélectionnés car ils fournissent empiriquement des informations discriminantes. Il existe beaucoup de tests différents pour chacun des nombreux pathogènes cibles. De plus, les techniques de biologie moléculaire (pour la caractérisation de gènes ou segments de gènes spécifiques) sont maintenant monnaie courante dans les laboratoires cliniques. Les méthodes taxonomiques modernes emploient souvent des méthodologies plus
complexes et s’appliquent à déterminer le profil de la composition structurale
des bactéries. Ceci implique souvent des approches basées sur la « biologie
moléculaire » ou la « chimie analytique ». Il est maintenant reconnu que
beaucoup de schémas classiques de différenciation des bactéries ne fournissent
qu’un aperçu limité de leurs relations génétiques et que certains cas sont
scientifiquement incorrects. De nouvelles données ont aboutit à renommer
certaines espèces bactériennes et dans certains cas ont nécessité une complète
réorganisation des relations au sein d’une même famille et entre de nombreuses
familles bactériennes. |
||||
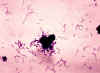
 Figure 1 Culture de Bacillus
anthracis sur des boîtes d’agar au sang et au bicarbonate. Colonies
lisses sur bicarbonate (gauche) et rugueuses sur l’agar au sang (droite).
CDC/Dr.
James Feeley
Figure 1 Culture de Bacillus
anthracis sur des boîtes d’agar au sang et au bicarbonate. Colonies
lisses sur bicarbonate (gauche) et rugueuses sur l’agar au sang (droite).
CDC/Dr.
James Feeley |
Termes taxonomiques (classification) Famille : un groupe de genres apparentés. Genre : un groupe d’espèces apparentées. Espèces : un groupe de souches apparentées. Type : un groupe de souches au sein d’une espèce (ex : biotypes, sérotypes). Le terme le plus communément utilisé est le nom d’espèce (ex : Streptococcus pyogenes - abréviation S. pyogenes). Il y a toujours deux parties dans le nom d’espèce, une définit le genre ici « Streptococcus » et l’autre l’espèce (ici « pyogenes »). Le nom de genre a toujours une majuscule mais pas le nom d’espèce. L’espèce et le genre sont sous-lignés ou en italique.
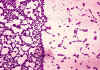
Etape 1. Des échantillons de fluides corporels (ex : sang, urine, liquide céphalo-rachidien) sont étalés sur des boîtes de culture et des colonies isolées de bactéries (visibles à l’œil nu) apparaissent après incubation d’un ou plusieurs jours (Figure 1). Chaque colonie est constituée de millions de cellules bactériennes. L’observation des colonies en terme de taille, texture, couleur et (si elles sont cultivées sur de l’agar au sang) de réaction d’hémolyse, est très importante dans la première étape de l’identification bactérienne. Le besoin en oxygène de la bactérie pour sa croissance est une autre caractéristique importante de différenciation. Etape 2. La coloration de Gram est effectuée sur les colonies et les cellules sont observées sous microscope. Etape 3. L’espèce des bactéries est déterminée en utilisant des colonies
isolées. Ceci requière souvent 24h de croissance supplémentaires. |
||||
|
Exemples de coloration de
Gram
|
La coloration de Gram Une colonie est séchée sur une lame et traitée comme suit (Figures 2 et 3) : Etape 1. Coloration au crystal violet. Etape 2. La fixation avec l’iodine stabilise la coloration au crystal violet. Toutes les bactéries restent violettes ou bleues. Etape 3. Extraction avec de l’alcool ou un autre solvant. Décoloration de certaines bactéries (Gram négatives) et pas d’autres (Gram positives). Etape 4. Contre-coloration avec la safranine. Les bactéries Gram positives sont déjà colorées avec le crystal violet et restent violettes. Les bactéries à Gram négatif sont colorées en rose. Sous le microscope l’apparence des bactéries est observée. Les questions à poser sont les suivantes :
Une autre colonie similaire provenant de la boîte d’isolation est ensuite examinée pour ses propriétés biochimiques ; par exemple, cette bactérie est-elle capable de fermenter un sucre tel que le lactose ? Dans certains cas, les bactéries sont identifiées (par exemple par agrégation) avec des anticorps commerciaux disponibles reconnaissant des antigènes de surface définis. D’autres testes moléculaires commerciaux sont maintenant largement utilisés.
Caractérisation taxonomique des bactéries Il existe une grande diversité même au sein d’une espèce. Ainsi les comparaisons d’espèces impliquent des comparaisons de multiples souches pour chaque espèce. Les comparaisons sont principalement basées sue des analyses chimiques et moléculaires.
Analyses chimiques Des outils sophistiqués sont disponibles pour étudier la composition structurale des bactéries (plus communément les acides gras, les sucres ou le profil des ubiquinones). La caractérisation de produits métaboliques secrétés (ex : alcools volatiles et les courtes chaines des acides gras) est aussi utile. Analyse moléculaire Il serait idéal de comparer les séquences de l’ADN chromosomique entier de bactéries, ce qui est possible mais encore très coûteux à l’heure actuelle. Des millions de nucléotides doivent être séquencés pour chaque souche. Il existe aujourd’hui des techniques très performantes de séquençage dit à « haut débit » permettant de séquencer plusieurs millions de bases en quelques heures. Cependant, même si ces techniques sont utilisées en recherche pour identifier et étudier les génomes des bactéries elles ne sont pas couramment utilisées en diagnostique. Alternativement, la similarité génomique a souvent été étudiée par le contenu en guanine (G) et cytosine (C), souvent exprimé en pourcentage (% GC). Ceci a été remplacé par deux alternatives –hybridation et séquençage (plus couramment du gène codant pour l’ARN 16S). L’homologie ADN-ADN (ou comment deux brins d’ADN de différentes bactéries se lient [s’hybrident] ensemble) est employé pour comparer la relation génétique de deux souches/espèces de bactéries. Si les ADN de deux souches bactériennes présentent un haut degré d’homologie (c'est-à-dire qu’ils se lient bien), les souches sont considérées comme des membres d’une même espèce. L’ADN de différentes espèces bactériennes (à moins d’être apparentées) ne présentent pas d’homologie. Depuis quelques années le séquençage des molécules d’ARN ribosomal 16S (ARNr 16S) est devenu le « standard » dans la taxonomie bactérienne. La molécule fait approximativement mille six cent nucléotides de long. La séquence de l’ARNr 16S fournit une mesure de la similarité génomique au-delà du niveau de l’espèce permettant une comparaison de la parenté dans tout le règne bactérien. Les espèces bactériennes fortement apparentées ont souvent des séquences d’ARNr identiques. La technique fournit ainsi des informations complémentaires à l’hybridation ADN-ADN. La détermination de la séquence des gènes de l’ARNr 16S et d’autres régions génétiques est utilisée pour l’identification dans les laboratoires de microbiologie clinique.
Approches pour un diagnostique rapide sans culture préalable Certains pathogènes humains (incluant les
agents responsables de la tuberculose, de la maladie de Lyme et de la
syphilis) soit ne peuvent pas être isolés en laboratoire ou poussent très
difficilement. Un isolement effectif peut être lent et dans certains cas
impossible. La détection directe de bactéries sans culture est possible dans
certains cas. Les séquences d’ADN bactérien peuvent être amplifiées directement à partir des fluides du corps humain (la PCR ou « Polymerase Chain Reaction »). De cette manière, de grandes quantités de gènes spécifiques ou des portions de gènes peuvent être générées et détectées directement. Par exemple, le diagnostique rapide de la tuberculose par cette technique est un grand succès. Enfin, l’observation microscopique directe de certains échantillons cliniques pour rechercher des bactéries peut fonctionner (ex : détection de M. tuberculosis dans les expectorations). L’identification sérologique de la réponse
immunitaire (recherche d’anticorps dans le sérum de patient) à un agent
infectieux peut fonctionner seulement à partir de plusieurs semaines après
l’infection. |
||||
|
|
|||||
|
|
|||||
|
ANIMATION |
|||||
|
TUTORIAL |
|||||
|
|
|
||||