| x | x | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| DOENÇAS INFECCIOSAS | BACTERIOLOGIA | IMUNOLOGIA | MICOLOGIA | PARASITOLOGIA | VIROLOGIA | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| INGLÊS |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ESPANHOL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ALBANES | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| PERSA | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ESCOLA DE MEDICINA DA UNIVERSIDADE DA CAROLINA DO SUL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Dê a
sua opinião CONTATO |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
APRENDA PLUGADO |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Palavras-Chave
Isolamento (cultura)
|
Identificação bacteriana no laboratório de diagnóstico versus taxonomia O isolamento e a identificação de bactérias de pacientes auxilia o tratamento uma vez que doenças infecciosas são causadas por bactérias diferentes apresentam uma variedade de cursos e conseqüências. Testar a susceptibilidade de isolados clínicos a antibióticos, ou seja, estabelecer a concentração inibitória mínima ou CIM, pode ajudar na seleção de antibióticos para a terapia. O reconhecimento de que certas espécies de bactérias (ou linhagens) estão sendo isoladas de forma atípica pode sugerir que tem ocorrido um surto da doença, por exemplo, de suprimentos hospitalares contaminados ou por não utilização de técnicas de assepsia adequadas por parte da equipe hospitalar.
Quando se suspeita de que pacientes estejam com uma infecção bacteriana, é usual proceder-se ao isolamento de colônias visíveis em cultura pura, em placas de meio de cultura sólido e, então, identificar o microrganismo. A identificação é baseada em princípios taxonômicos aplicados à situação microbiológica em curso. No laboratório de diagnóstico, muitas amostras devem ser caracterizadas por dia e os resultados obtidos o mais rápido possível. Os testes devem ser facilmente aprendidos, de baixo custo e poderem ser realizados rapidamente.
Os métodos clássicos para a identificação de bactérias são baseados em características morfológicas e bioquímicas. Têm sido desenvolvidos testes de diagnóstico provêm informações discriminatórias. Há numerosos testes para cada um dos muitos patógenos. Técnicas de biologia molecular (para a caracterização de genes específicos ou segmentos de genes) estão se tornando comuns nos laboratórios de diagnóstico.
Abordagens taxonômicas modernas empregam metodologias mais complexas e centram-se no estabelecimento da composição estrutural das bactérias. Isto freqüentemente envolve abordagens baseadas na biologia molecular ou na química analítica. Atualmente se reconhece que muitos esquemas clássicos de distinção entre bactérias provêm pouco conhecimento sobre suas relações genéticas e, em alguns casos, estão cientificamente incorretos. Novas informações têm acarretado a alteração dos nomes de certas espécies bacterianas e, em certas situações, requereu-se a total reorganização das relações dentro e entre muitas famílias bacterianas.
Termos taxonômicos (classificação) Família: um grupo de gêneros relacionados. Gênero: um grupo de espécies relacionadas. Espécies: um grupo de linhagens relacionadas. Tipo: um conjunto de linhagens dentro de uma espécie (biótipo, sorotipo). Linhagem: um isolado específico dentro de uma espécie particular. O termo mais comumente usado é o nome da espécie, por exemplo, Streptococcus pyogenes - abreviação: S. pyogenes. O nome da espécie é sempre composto por duas palavras, a primeira define o gênero, no caso, Streptococcus, e a segunda a espécie, neste caso, pyogenes. A primeira letra do nome do gênero é sempre maiúscula, mas o nome da espécie é todo em minúsculas. Ambos os nomes são italizados ou grifados.
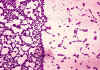
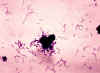
Passos no isolamento e identificação de uma bactéria Passo 1: amostras de fluidos orgânicos (por exemplo, sangue, urina, fluido cefalorraquidiano) são espalhados em placas contendo meio de cultura sólido; colônias isoladas de bactérias (visíveis a olho nu) aparecem após a incubação das placas por um a vários dias (Figura 1). Cada colônia consiste de milhões de células de bactérias. A observação das características dessas colônias em relação a tamanho, forma, textura, cor e (se crescidas em meio agar sangue) reações de hemólise é altamente importante como um primeiro passo na identificação bacteriana. Se a bactéria requer ou não oxigênio para crescer é uma outra importante característica para a identificação. Passo 2: as colônias isoladas são coradas pelo método de Gram e células individuais de bactérias são observadas ao microscópio. Passo 3: As bactérias são identificadas usando-se colônias isoladas. Isto freqüentemente requer outras 24 horas de incubação adicional.
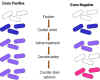
A coloração de Gram A colônia é dessecada sobre uma lâmina e tratada como segue (Figuras 2 e 3) Passo 1: coloração com cristal violeta Passo 2: fixação com lugol estabiliza a coloração com o cristal violeta. Todas as bactérias se coram em azul ou púrpura. Passo 3: extração com álcool ou outro solvente. Descora algumas bactérias (Gram negativas) e não outras (Gram positivas). Passo 4: Coloração secundária com safranina. As bactérias Gram positivas já estão coradas com o cristal violeta e permanecem azuis. As bactérias Gram negativas se coram em rosa. Observa-se a aparência das bactérias sob microscópio. Pergunta-se:
Além da coloração de Gram, há outros métodos de coloração menos comumente empregados, por exemplo, a coloração de esporos ou cápsulas. Uma outra colônia obtida a partir daquela primariamente isolada é, então, testada quanto às suas propriedades bioquímicas. Por exemplo, a bactéria fermenta algum açúcar, tal como a lactose? Em alguns casos, as bactérias são identificadas (por exemplo, por aglutinação) com anticorpos disponíveis comercialmente e que reconhecem antígenos de superfície específicos. Outros testes moleculares comerciais são agora amplamente utilizados.
Caracterização Taxonômica das Bactérias Há considerável diversidade mesmo dentro de uma espécie. Portanto, comparações entre espécies envolvem comparações de múltiplas linhagens de cada espécie. Essas comparações são primariamente baseadas em análises químicas e moleculares.
Análises químicas Existe uma tecnologia sofisticada para o estudo da composição estrutural das bactérias (mais comumente, a caracterização de ácidos graxos, carboidratos ou ubiquinona). A caracterização de produtos metabólicos secretados, como por exemplo, álcoois voláteis e ácidos graxos de cadeia curta, são também empregados.
Análises moleculares Seria ideal a comparação de seqüências completas de cromossomos bacterianos, mas isto não é possível atualmente. Milhões de nucleotídios têm que ser seqüenciados para cada linhagem. Nos últimos anos, conseguiu-se seqüenciar os genomas completos de tipos representativos (ou seja, uma linhagens) de umas poucas espécies bacterianas. Em cada caso, isto envolveu uma quantidade enorme de trabalho por grandes grupos de pesquisa dedicados ao seqüenciamento de genomas bacterianos. Alternativamente, as similaridades genômicas entre grupos bacterianos têm sido medidas pelos conteúdos de guanina (G) mais citosina (C), usualmente expressa como uma porcentagem (%GC). Isto foi substituído por outras duas alternativas: hibridização e seqüenciamento (mais comumente do gene codificando o RNAr 16S).
A homologia DNA-DNA (ou seja, o quanto duas cadeias de DNA de diferentes bactérias podem se hibridizar) é empregada para comparar o grau de relacionamento genético entre espécies ou linhagens de bactérias. Se os DNAs de duas linhagens bacterianas apresentam alto grau de homologia essas linhagens são consideradas como membros da mesma espécie. Os DNAs de diferentes espécies bacterianas (a menos que sejam estreitamente relacionadas) não apresentam homologia.
Nos últimos anos, o seqüenciamento de moléculas de RNA da subunidade ribossômica 16S (RNAr 16S) tornou-se o "padrão de ouro" na taxonomia bacteriana. Essa molécula tem, aproximadamente, dezesseis mil nucleotídios de comprimento. A seqüência do RNAr 16S provê uma medida da similaridade genômica acima do nível de espécie permitindo comparações de relacionamentos genéticos por todo o reino bacteriano.
Espécies bacterianas estreitamente relacionadas frequentemente têm seqüências RNAr 16S idênticas. Essa técnica, portanto, fornece informações complementares à hibridização DNA-DNA. Determinações de seqüências dos genes para o RNAr 16S e de outras regiões genéticas são usadas na identificação no laboratório de microbiologia clínica.
Abordagens para diagnóstico rápido sem cultura prévia Certos patógenos humanos (incluindo os agentes causadores da tuberculose, doença de Lyme e sífilis) ou não podem ser isolados em laboratório ou crescem muito pobremente em cultura. Um isolamento bem sucedido pode ser demorado e em alguns casos até mesmo impossível. A detecção direta de bactérias sem cultivo é possível em alguns casos. Uma abordagem simples para diagnóstico rápido - a detecção de antígenos bacterianos - é usada em muitos consultórios médicos para a detecção de Streptococcus do grupo A. Um esfregaço é feito na garganta do paciente pela técnica do Swab e antígenos estreptocócicos extraídos diretamente da zaragatoa (sem cultura bacteriana prévia) são detectados por aglutinação de esférulas recobertas por anticorpos. Seqüências de DNA bacteriano podem ser amplificadas diretamente de fluidos corporais humanos (pela reação em cadeia da polimerase, PCR). Por esta técnica, grandes quantidades de genes específicos ou porções de genes podem ser geradas e rapidamente detectadas. Por exemplo, o diagnóstico rápido da tuberculose tem sido obtido grandemente bem sucedido. Finalmente, a observação microscópica direta de certos espécimes clínicos pela presença de bactérias pode ser muito útil, como no caso da detecção de M. tuberculosis em escarro. A identificação sorológica de uma resposta a anticorpos do agente infeccioso (presentes no escarro do paciente) só pode ser bem sucedida várias semanas após a ocorrência da infecção.
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||