|
x |
x |
|
 |
 |
|
INFECTIOUS
DISEASE |
BACTERIOLOGY |
IMMUNOLOGY |
MYCOLOGY |
PARASITOLOGY |
VIROLOGY |
|
|
VIROLOGIA – CAPÍTULO VINTE E CINCO
CORONA VÍRUS:
COLDS,
SARS, MERS E COVID-19
Dr Richard
Hunt
Professor
Department of Pathology, Microbiology and Immunology
University of South Carolina School of Medicine
Columbia
South Carolina
Tradução:
PhD. Myres Hopkins
|
|
ENGLISH |
|
|
|
|
Você poderá gostar de ler também sobre:
English
Vaccines
Pandemics
Social Distancing and Masks
Portugues
Pandemias
Por que usar uma máscara em uma pandemia?
|
|
|
|
|
|
Logo image © Jeffrey
Nelson, Rush University, Chicago, Illinois and
The MicrobeLibrary |
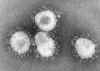
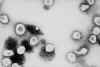 Figura 1
Figura 1
Coronavírus é um grupo de vírus que têm um halo com aparência de coroa
quando visto ao microscópio
CDC/Dr. Fred Murphy (top) CDC/Dr. Erskine Palmer (bottom)
|
INTRODUÇÃO
Coronavírus (figura 1) são os maiores vírus de RNA e têm cerca de 100nm
de diâmetro. Eles infectam humanos e animais nos quais eles causam
doença respiratória e entérica.
A ordem Nidovirales de vírus que infecta vertebrados (incluindo
humanos) e hospedeiros invertebrados consiste em quatro famílias:
-
Coronaviridae.
Há duas famílias - Coronavirinae (Coronavírus), que infecta
mamíferos e aves e a Torovirinae (Torovírus) (figura 2) que
infecta vertebrados, especialmente gado, porcos e cavalos.
-
Arteriviridae que infecta vertebrados, mamíferos em maioria,
incluindo o vírus da arterite equina.
-
Roniviridae
que infecta crustáceos.
-
Mesoniviridae
que infecta insetos.
Eles são vírus de RNA grandes, de fita única de sentido positivo
(sentido do RNAm) , envelopados e há sete, todos coronavírus que são
conhecidos por causar doença humana.
Este
capítulo irá discutir apenas os Coronavírus, visto que eles são
particularmente importantes em doença respiratória humana, causando
cerca de um terço dos “resfriados comuns” e as recentemente reconhecidas
síndromes respiratórias agudas graves: SARS, MERS e COVID-19.
|
 Figura 2
Torovírus
Figura 2
Torovírus
© Queen's University, Belfast
|
Coronavíruses
Corona =
Latin: crown, κορώνη da Grécia Antiga – pelo fato de se parecer com
coroas-de-espinhos em uma microscopia eletrônica.
Os Coronavírus
são os mais importantes da Ordem Nidovirales em doença humana e são divididos em
quatro gêneros:
Alfa-coronavírus, que inclui HCoV-NL63 e HCoV-229E que normalmente causa
infecções amenas do trato respiratório superior em humanos. O vírus da
diarréia epidêmica suina é também um membro desta classe de coronavírus.
Beta-coronavírus, que inclui MERS CoV, SARS CoV-1 e SARS CoV-2 (que causa a
doença COVID-19) e HCoV-OC43 e HCoV-HKU1 (que normalmente causa sintomas
respiratórios menos severos)
Gama-coronavírus, que infecta principalmente aves.
Delta-coronavírus, que também infecta principalmente aves, embora o
coronavírus delta suino seja importante na pecuária. Ele emergiu em 2014 e
pode ter partido de um hospedeiro aviário.
Três
β-coronavírus têm causado epidemias de pneumonia mortal em humanos desde o
início deste século. A síndrome respiratória aguda grave (SARS-CoV-1) (do inglês
Severe Acute Respiratory Syndrome Coronavirus) foi inicialmente relatada em
2002. A doença que ele provoca, a síndrome respiratoria aguda grave (SARS) é
altamente infecciosa e tem um alto índice de fatalidade de 10%. Seu epicentro
foi a China e Hong Kong, mas foi amplamente contida em 2003, embora alguns casos
adicionais ocorreram em 2004.
A síndrome
respiratória do oriente médio pelo Coronavírus MERS-CoV (do inglês Middle East
Respiratory Syndrome Coronavirus) emergiu na Arábia Saudita e outros países da
Península Arábica em 2012. A síndrome Respiratoria do Oriente Médio (MERS) tem
um índice de fatalidade muito alto em humanos de cerca de 35%. Há também casos
de MERS associados a viagens na Europa, Ásia e América do Norte. Em maio de 2014
o CDC relatou dois casos importantes de MERS nos Estados Unidos em pessoas que
haviam viajado da Arábia Saudita.
SARS CoV-2 é o
agente causador da Doença do Corona Vírus 2019 (COVID-19) que abarcou o globo em
uma doença pandêmica em 2020. Assim como na SARS, o epicentro da COVID-19 foi a
China (Wuhan, a capital da Província de Hubei). Foi provada altamente infecciosa
e tem um índice de fatalidade em torno de 2%, embora isso possa mudar à medida
que novos dados se tornem disponíveis. A mortalidade ocorre mais
proeminentemente entre os idosos com complicações co-mórbidas, mas pessoas de
todas as idades têm morrido por causa da doença.
Todos os três
coronavírus são vírus zoonóticos (provém de animais) que cruzam barreiras de
espécies. Morcegos são os hospedeiros animais primários para o MERS CoV e SARS
CoV-1 e -2. SARS CoV-1 pode ter passado de morcegos para humanos via os chamados
musang ou civeta de palmeira asiática, enquanto o SARS CoV-2 pode ter infectado
humanos a partir de morcegos via pangolins, também animais asiáticos. Em ambos
os casos, o vírus provavelmente infectou humanos que tiveram contato com animais
selvagens em mercados de animais selvagens vivos na China. Os hospedeiros
intermediários do MERS CoV são camelos dromedários.
Vários outros
coronavírus zoonóticos são endêmicos na pupulação humana. Eles causam cerca de
um terço das infecções do trato respiratório como rinorréia (nariz escorrendo),
dor de cabeça, espirros, moleza e dor de garganta ganta. Corisa (inflamação
aguda do trato respiratório superior) com febre e tosse é vista em 10 a 20% dos
casos. Em alguns casos, estas consideradas infecções amenas podem levar a
complicações severas ou morte em crianças pequenas, nos idosos e indivíduos
imunocomprometidos. Esses vírus que também podem causar diarréia são HCoV-NL63 e
HCoV-229E (alfa), HCoV-OC43 e HCoV-HKU1 (beta).
|
| |
CORONAVÍRUS “RESFRIADOS”
Em humanos, cerca de um
terço dos resfriados são causados pelos coronavírus, sendo o
principal local de replicação dos coronavírus as células epiteliais
do trato respiratório. Os sintomas são semelhantes ao dos resfriados
por rinovírus com um período de incubação de cerca de 3 dias. A
disseminação viral é limitada pela resposta imune de muitos
pacientes, mas esta imunidade tem vida curta. Os sintomas podem
durar cerca de uma semana com variações consideráveis entre os
pacientes. Muitas vezes não há sintoma aparente, mas o paciente
ainda elimina o vírus infeccioso.
Embora infecções por
coronavírus sejam normalmente localizadas no trato respiratório
superior, elas podem se espalhar por outros órgãos. Em humanos,
esses vírus têm sido implicados em infecções do ouvido médio, em
algumas pneumonias em pacientes imunocomprometidos, em miocardites,
mas em animais infecções sistêmicas podem ser muito mais severas
(ex. peritonite infecciosa felina).
Diferentemente dos rinovírus
(Picornaviridae) que não são envelopados, os coronavírus são
muito mais instáveis. A transmissão é por transferência da secreção
nasal, tais como em aerossóis causados por espirros. Vírus que
infectam células epiteliais do trato entérico causam diarréia. Isto
pode ocorrer em neonatos humanos, mas é comum em muitos animais
jovens onde a infecção pode ser fatal.
Epidemiologia
A maioria das pessoas tem anticorpos
anti-coronavírus. Mas a reinfecção é comum, indicando que há muitos
sorotipos do vírus circulando na população humana. E como ocorre na
maioria das infecções respiratórias, resfriados causados por
coronavírus são mais comuns no inverno por causa do contato próximo
e humidade baixa. Os principais surtos ocorrem a cada poucos anos
com um ciclo que depende do tipo de vírus envolvido.
Diagnóstico
A maioria das infecções pelo
coronavírus não são diagnosticadas e a doença é auto-limitada (cura
por si mesma). O diagnóstico pode ser feito usando
imuno-eletro-microscopia e sorologia. Não há antivirais para
infecções rotineiras por coronavírus, mas medicamentos sem
prescrição para alíviar os sintomas são úteis.
VÍRUS DA
HEPATITE MURINA E ESCLEROSE MÚLTIPLA
Um fato interessante é que uma
linhagem neurotrópica do vírus da hepatite murina (um beta coronavírus)
pode causar uma doença em roedores que se parece muito com a esclerose
múltipla, levando à sugestão de seu envolvimento na doença humana;
desmielinização, uma característica da esclerose múltipla em
modelo-animal roedor, está ligada à proteína S e tem sido sugerido que a
doença resulte de mimetismo molecular no qual uma resposta imune à
proteína S leve a um ataque imune à mielina. Entretanto, embora o vírus
possa ser detectado no cérebro de pacientes, o link para esclerose
múltipla continua sem prova.
|
|
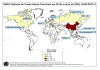 Figura
3
Figura
3
Mapa de casos prováveis de SARS. 02 de junho de 2003
OMS
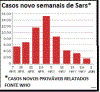 Figura 4
Figura 4
.Novos casos semanais de SARS
©
WHO/BBC
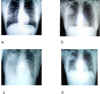 Figura 8.
Figura 8.
Radiografia de tórax de paciente típico com
síndrome respiratória aguda grave (SARS). a, dia 5 da
sintomatologia; b, dia 10; c, dia 13; d, dia 15.
Li-Yang Hsu, Cheng-Chuan Lee, Justin A. Green, Brenda Ang, Nicholas I.
Paton, Lawrence Lee, Jorge S. Villacian, Poh-Lian Lim, Arul Earnest, and
Yee-Sin Leo - Tan Tock Seng Hospital, Tan Tock Seng, Singapore.
Doenças
Infecciosas e Emergentes
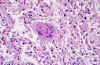 Figura 9 Mudanças citoarquiteturais patológicas indicativas de dano
alveolar difuso, além de uma célula gigante multinucleada com ausência
de inclusões virais conspícuas.
Figura 9 Mudanças citoarquiteturais patológicas indicativas de dano
alveolar difuso, além de uma célula gigante multinucleada com ausência
de inclusões virais conspícuas.
CDC/Dr. Sherif Zaki
|
SÍNDROME
RESPIRATÓRIA AGUDA GRAVE (SARS)
No fim do ano 2002 uma nova
síndrome foi observada no sudeste da China (Província de Guangdong). Foi
denominada de Síndrome Respiratória Aguda Grave (SARS). Esta doença, que
já foi relatada na Asia, América do Norte e Europa (figura 3), é
caracterizada por uma febre de mais de 38 graus (100.4 em graus
Fahrenheit) acompanhada de dor de cabeça, mal estar geral e dores. Os
sintomas respiratórios são inicialmente e normalmente amenos, mas após
alguns dias a uma semana o paciente pode desenvolver uma tosse não
produtiva e a respiração difícil (dispnéia). O stress respiratório leva
à morte em 3-30% dos casos. Testes laboratoriais mostram uma redução no
número de linfócitos e um aumento da atividade da aminotransferase, o
que indica dano hepático.
A epidemia inicial da SARS
chegou a um pico em abril de 2003 e em junho começou a cair (figura 4).
Nessa época houve cerca de 8.000 casos no mundo inteiro com 775 mortes.
Além disso, houve uma perda econômica de bilhões de dólares.
Vírus de pacientes infectados
foram crescidos em células Vero E6 de macacos em cultura de tecidos e
identificados como um novo coronavírus (SARS-CoV). Ele tem um genoma de
29,727 bases e onze fases de leitura abertas. A organização do genoma é
muito similar à de outros coronavírus (5’ replicase (rep), espícula (S),
envelope (E), membrana (M), nucleocapsídio (N)-3′
e pequenas regiões não traduzidas em ambos os terminais). O gene da
replicase (RNA polimerase) ocupa dois terços da ponta 5’ do genoma e
tem, assim como outros coronavírus, duas fases de leutura abertas
sobrepostas. Ele também codifica para uma protease que é parte da
poliproteína RNA polimerase. Há nove fases de leitura aberta possíveis
que não são encontradas em outros coronavírus e podem codificar para
proteínas que são típicas do vírus SARS. Usando testes com anticorpos,
coronavírus-SARS têm sido associados com casos de SARS através do mundo.
Diagnóstico
O Centro de Controle de Doenças dos
Estados Unidos recomenda uma radiografia de tórax (figura 8),
oximetria de pulso (um teste usado para medir a saturação de
oxigênio do sangue), culturas de sangue, coloração do escarro pelo
Gram e cultura, além de teste para outros patógenos respiratórios
virais, notadamente influenza A e B e vírus respiratório sincicial.
Um espécime para teste de Legionella e antígeno pneumocócico
urinário poderia ser considerado. Pessoas com suspeita de SARS devem
ser isoladas e submetidas a quarentena.
A infecção por SARS-CoV-1
mostra alterações indicativas de dano alveolar difuso, assim como
uma célula gigante multinucleada sem inclusões virais conspícuas
(figura 9).
Tratamento
Não há
tratamento acordado contra a SARS além do gerenciamento dos
sintomas. As drogas estão em desenvolvimento e são de particular
interesse as que possam bloquear a função da protease, visto que
isso é crucial para o vírus. Não há vacina aprovada contra o vírus
SARS, embora algumas tenham sido desenvolvidas. Existem programas de
vacinação veterinária com sucesso modesto contra vários coronavírus
de importância econômica.
|
| |
SÍNDROME
RESPIRATÓRIA DO ORIENTE MÉDIO
Em 2012 uma doença causada por
um Coronavírus novo apareceu no Oriente Médio, mais precisamente na
Arábia Saudita. Inicialmente, todos os pacientes moravam ou teriam
visitado o Oriente Médio, embora alguns tenham subsequentemente viajado
para a Europa onde apareceram casos na França, no Reino Unido e na
Alemanha. Após a infecção inicial, o vírus se espalha para contatos
próximos indicando uma transmissão humano a humano. Os pacientes
desenvolveram pneumonia e às vezes insuficiência renal com taxa de
fatalidade de até 50%, embora esta alta fatalidade possa refletir a
falha no diagnóstico de casos menos virulentos. O vírus foi
inicialmente chamado de Novo Corona Vírus (nCoV) e depois chamado de
“Coronavírus da Síndrome Respiratória do Oriente Médio” (em inglês
(MERS-CoV) e é distindo do Coronavírus SARS. Ele pode ser tratado com
interferon α2b e ribavirina.
Mais uma vez
a origem deste novo coronavírus é morcegos, especificamente o morcêgo
pipistrelle (morcêgo anão), sendo o hospedeiro intermediário os camelos
dromedários. Aparentemente, por análise filogenética, o vírus entrou na
população humana em torno do ano 2011 |
| |
A DOENÇA DO
CORONAVÍRUS 2019 (COVID-19)
Em 31 de Dezembro de 2019, houve
um caso de pneumonia de etiologia desconhecida admitida em hospital de
Wuhan, China. Foi rapidamente estabelecido que não era nem SARS nem MERS
e nem outro patógeno respiratório; outros casos se sucederam. Eles
tinham as características de uma infecção por coronavírus e rapidamente
foi demonstrado serem resultantes de um coronavírus até então
desconhecido, e que foi inicialmente chamado de 2019-nCoV, mas agora foi
renomeado para SARS-Coronavírus 2 (SARS-CoV-2), pois ele também provoca
uma síndrome respiratória aguda grave.
Em 11 de Janeiro havia 41 casos
desta doença em Wuhan; a maioria era benigna, mas sete delas foram
infecções sérias e naquela época havia uma morte. A doença se espalhou
via transmissão humano para humano, foi altamente infecciosa e logo foi
encontrada fora de Wuhan, nas vizinhanças da Província de Hubei e depois
em outros lugares da China. O primeiro caso fora da China foi na
Tailândia e a paciente (uma mulher de 61 anos) tinha vindo de Wuhan.
Após severas restrições de movimento em Wuhan e nas vizinhanças da
Província de Hubei, o govêrno Chinês conseguiu controlar a epidemia, mas
a COVID-19 se espalhou pelo mondo inteiro. Ao final da primeira onda de
infecções pelo SARS CoV-2 (Abril de 2020), a China relatou 81.865 casos,
3.335 mortes e 77.370 pacientes recuperados. De um modo geral a taxa de
fatalidade de caso em Wuhan foi de 0.9-2.1%.
Ao final da segunda semana de
Abril de 2020, houve 1.5 milhões de casos de COVID-19 no mundo inteiro
com 91.000 mortes e 342.000 pessoas recuperadas.
Sintomas
da COVID-19
Dois a quatorze dias após a
exposição, de acordo com o CDC, os sintomas iniciais são:
-
Febre
-
Tosse
-
Falta de ar (dispnéia)
-
Fadiga
-
Dor muscular e no corpo
-
Dor de cabeça
-
Congestão
-
Náusea
-
Vômitos
-
Diarréia
-
Muitas vezes perda do olfato e paladar
Subsequentemente, sintomas
mais severos podem aparecer, incluindo:
-
Respiração problemática
-
Dor persistente ou pressão no peito
-
Confusão ou incapacidade de despertar
-
Lábios ou face azulados
Em algumas crianças há
sintomas semelhantes à Síndrome de Kawasaki.
Entretanto, muitas pessoas
infectadas não apresentam sintomas ou apresentam sintomas amenos,
como tosse e febre.
Mortalidade
SARS e MERS têm um alto índice de mortalidade com aproximadamente
10% de pacientes de SARS e aproximadamente 37% de pacientes de MERS
que sucumbem à doença. COVID-19 aparenta ter uma taxa de mortalidade
muito mais baixa de cerca de 2%, embora esta aumenta com a idade do
paciente e a presença de complicações subjacentes.
Idade Taxa de Mortalidade (%)
·
80+
14
·
70-79 8
·
60-69 3.6
·
50-59 1.3
·
40-40 0.4
·
30-39 0.2
·
20-29 0.2
·
10-19 0.2
Homens têm uma taxa de fatalidade maior do
que mulheres
Mulheres 1.7
Homens 2.8
Certas comorbidades aumentam
muito a taxa de morte, incluindo doença cardíaca, diabetes,
hipertensão e doença respiratória crônica.
Transmissibilidade
Como em março de 2020, a
transmissibilidade (fator de reprodução R0) do SARS-CoV-2 aparentou
ser em torno de 2 a 3. Isso significa que cada pessoa infectada
espalhou o vírus para uma média de 2 a 3 outras pessoas.
|
|
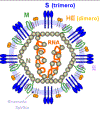 Figura
10
Figura
10
Estrutura do coronavírus structure.
Adaptado de Lai e Homes. In Fields'
Virology. Lippencott
 Figura 11
Figura 11
Morfologia ultraestrutural de um coronavírus visto por microscopia
eletrônica. As espículas (proteína S) dão a aparência de uma coroa, daí
o nome. As proteínas E e M estão também localizadas na superfície
externa do vírus.
CDC
 Figura 12
Figura 12
Genoma do SARS-CoV-2 mostrando a ordem dos genes e as proteínas por eles
codificadas.
 Figura
13 RNAs mensageiros dos coronavírus. É formado um conjunto localizado de
RNAs com uma extremidade 3’ em comum. O RNAm da polimerase (pol) tem o
mesmo comprimento do RNA genômico. Os restantes são truncados na
extremidade 5’, embora todos tenham uma sequência líder comum. Figura
13 RNAs mensageiros dos coronavírus. É formado um conjunto localizado de
RNAs com uma extremidade 3’ em comum. O RNAm da polimerase (pol) tem o
mesmo comprimento do RNA genômico. Os restantes são truncados na
extremidade 5’, embora todos tenham uma sequência líder comum. |
ESTRUTURA DO
CORONAVÍRUS
Os coronavírus são vírus de fita
simples com sentido positivo. Diferentemente do usual para vírus de RNA,
eles são muito grandes, com um genoma de cerca de 30kb. Este tamanho tão
longo traz consequências na sua taxa de mutação, o que será discutido
abaixo. O fato de estes vírus serem de sentido positivo, significa que
seu genoma éstá no mesmo sentido do RNAm e o RNA genômico poder ser
usado como um RNAm assim que a célula for infectada. Partículas virais
em maturação brotam através das membranas intracelulares e ganham um
envelope lipídico (ou seja, coronavírus são vírus envelopados). Isso
traz consequências para o controle da infecção, visto que eles são
provavelmente de menor estabilidade do que os vírus não envelopados e
serem mais sensíveis a detergentes e solventes orgânicos. A estrutura de
um coronavírus é mostrada na figura 10 e a morfologia externa na figura
11.
Genoma do
vírus da
COVID-19
O vírus SARS-CoV-2 tem um genoma
de 29.829 bases e é como um RNAm típico (figura 12). Ele tem uma cauda
3’ poli A e é capeado no terminal 5’. Este último consiste em um
nucleotídeo de guanina ligado ao RNAm via uma ligação trifosfato não
usual de 5’ para 5’. Esta guanosina é metilada na posição 7 por uma
metiltransferase codificada pelo vírus. Além disso, a extremidade 5’ é
metilada nos grupos hidroxílicos 2’ dos dois primeiros resíduos de
ribose. Este cap proporciona resistência a degradação por exonucleases
5’ na célula.
Cerca de dois terços do genoma,
começando pelo terminal 5’, codifica para a proteína não-estrutural
(NSP) 1ab. Esta região é também conhecida como o gene da replicase,
embora outras proteínas além da replicase (RNA polymerase) sejam
codificadas neste gene. Proteínas não estruturais são proteínas
codificadas pelo vírus que não são parte da partícula viral madura, mas
são usadas na replicação e maturação do vírus. As sequências
codificadoras para as proteinas 1a e 1b não estão na mesma fase de
leitura, mas o ribossomo realiza um pulo de fase a -1 ao final do gene
da proteína 1a de forma que é produzida uma longa poliproteína. Esta é
então clivada por uma protease codificada pelo vírus para fazer as
proteínas 1a e 1b. As proteínas 1a e 1b são por si sós poliproteínas e
são clivadas pela atividade proteásica de 16 proteínas menores não
estruturais codificadas pelo vírus. Proteínas não estruturais são
necessárias antes das proteínas estruturais, visto que elas estão
envolvidas na síntese do RNA viral e proteínas, e assim precisam ser
produzidas logo após a infecção da célula. Isso é feito pela tradução do
RNA de fita positiva que entra, que tem todas as características de um
RNAm celular.
Os genes para as proteínas
estruturais estão localizados no terço 3’ do genoma. Estes são
transcritos a um conjunto de fitas de RNA negativas complementares, que
são moldes (templates) para a transcrição de um conjunto localizado de
RNAm sub-genômicos a partir dos quais as proteínas estruturais são
traduzidas (figura 13).
No SARS CoV-1 e -2 e MERS CoV,
há quatro fases de leitura abertas nesta região 3’ que codificam para:
·
A proteína da espícula (S) pela qual o
vírus se encaixa no receptor da célula hospedeira
·
A proteína de membrana (M)
·
A proteína do envelope (E)
·
A proteína do nucleocapsídeo (N)
Além disso, há uma quantidade de
fases de leitura abertas menores que codificam para proteínas que podem
ser estruturais ou podem assumir alguma função acessória.
Proteínas
do Coronavírus
As
proteínas não estruturais NSP 1 – NSP16
A função de algumas dessas
proteínas é desconhecida, mas algumas estão envolvidas no controle
do metabolismo do ácido nuclêico das células infectadas. Na maioria
dos casos isso é inferido a partir de pesquisas sobre outros
coronavírus, tais como os estreitamente relacionados SARS CoV-1 e
MERS CoV, não sendo, portanto, resultado de investigações sobre o
SARS-CoV-2.
NSP1 – Redução da síntese de proteínas da célula hospedeira
A NSP1 desliga a tradução a
proteínas pela célula hospedeira muito eficientemente ao ligar-se às
sub-unidades 40S dos ribossomos. O complexo formado pela NSP1 e a
subunidade ribossomal também age como uma enzima que inibe a
tradução de proteínas pela célula hospedeira por meio de uma
clivagem endonucleolítica próximo à 5'UTR (região não traduzida) do
RNAm hospedeiro, levando à degradação do RNAm da célula hospedeira.
Isso suscita a pergunta de que por que os RNAm virais não são
clivados de maneira similar, visto que, para todos os efeitos eles
são como os RNAm celulares? Acontece que NSP1 se liga a uma
estrutura em grampo no 5'UTR do RNA do SARS CoV-1 e esta interação
estabiliza o RNAm que carrega o grampo específico e aumenta a
tradução de proteínas virais.
NSP2 – Uma proteína de função desconhecida
A NSP2 expressada em células
usando a transdução retroviral foi especificamente recrutada para
replicações virais complexas. Ela não é requisitada para replicação
viral em células em cultura, embora a deleção das sequências
codificadoras de NSP2 atenuem o crescimento viral e a síntese de
RNA. Fora isso, a função da NSP2 não é conhecida.
NSP3 – Uma proteína multifuncional que contém uma protease
Esta é a maior proteína codificada pelo genoma do coronavírus, com
um tamanho de cerca de 200 kD. É um componente essencial do complexo
replicação/transcrição do vírus e atravessa a membrana do retículo
endoplasmático. E como se espera de uma proteína tão grande, ela tem
vários domínios com diferentes funções:
Um domínio tipo ubiquitina 1 (Ubl1). Liga-se ao
RNA fita simples e interage com a proteína (N) do nucleocapsídio. É
essencial para a replicação viral a qual cessa quando o domínio UbL1
é parcialmente deletado.
Um domínio rico em ácido glutâmico (também
chamado de “região hipervariável”)
Um domínio tipo protease (papaína) (protease 1
(PLpro). Este libera NSP1, NSP2 e
NSP3 da região N-terminal das poliproteínas 1a e 1ab.
Um macrodomínio ou
domínio X. Este não é necessário para a replicação do RNA, mas pode
estar envolvido na neutralização da resposta imune inata do
hospedeiro.
Outro domínio tipo ubiquitina 2 (Ubl2). A função
desta não é conhecida.
Outro domínio tipo protease (papaina) (protease 2
(PL2pro)).
NSP3 ectodomínio (3Ecto, “domínio zinc-finger”).
Este é o único domínio localizado no lado luminal do retículo
endoplasmático em SARS-CoV-1 NSP3.
Acredita-se que ele liga íons metálicos e contém um oligosacarídeo
ligado a asparagina.
Domínio Y1, a função deste não é conhecida.
Domínio CoV-Y, que também tem função não
conhecida. Os domínios Y1 e CoV-Y estão no lado citosólico do
retículo endoplasmático.
Há dois domínios
transmembrana em NSP3 que aparentam cruzar a membrana do retículo
endoplasmático duas vezes. Estes, mais o domínio 3Ecto são
importantes para o domínio protease PL2pro clivar o sítio entre NSP3
e NSP4 no SARS-CoV-1. O domínio transmembrana pode aproximar PL2pro
do sítio de clivagem entre as proteínas associadas a membrana NSP3 e
NSP4.
NSP3 junto com NSP4 e NSP6
são necessárias para a formação das vesículas de dupla membrana que
são características das células infectadas por coronavírus.
NSP4
– Reorganização de membranas celulares
NSP4 é uma glicoproteína que
atravessa a membrana do retículo endoplasmático quatro vezes com
três regiões de alça. Alças 1 e 3 são expostas ao lúmen do retículo
endoplasmático, enquanto a alça 2 e os terminais N e C são
citosólicos. Há dois sítios de glicosilação ligados a asparagina na
alça 1.
Como será descrito abaixo,
muitos vírus de RNA de fita positiva, incluindo coronavírus,
modificam as membranas citoplasmáticas da célula hospedeira que são
sítios de síntese de RNA viral e da formação de complexos de
replicação viral. Coronavírus induzem vesículas de dupla membrana e
quando células infectadas são analisadas por miscroscopia
eletrônica, mutantes NSP4 têm morfologia aberrante nas suas
vesículas de dupla membrana quando comparados com células infectadas
com o vírus tipo selvagem. Portanto, NSP4 pode ter um papel de
organização das membranas das vesículas, o que é importante em uma
síntese de RNA e replicação viral, embora seus papéis nesses
processos não estejam claro. Um sítio de glicosilação no lado
luminal parece ser importante na síntese de RNA.
NSP5 - Uma protease
NSP5 (3CLpro, Mpro) é uma
protease que cliva outras proteínas NSP em sítios de clivagem 11 e é
essencial para a replicação do vírus. A protease NSP5 é também uma
antagonista de interferon que inibe a produção de interferon-beta
induzida pelo vírus Sendai em células infectadas pelo direcionamento
ao mirar uma proteína chamada modulador essencial (NEMO) NF-κB.
NSP6 – Reorganização das membranas celulares
NSP6 está também envolvida
na formação da dupla membrana no interior da célula infectada. Ela
induz vesículas perinucleares localizadas ao redor do centro de
organização dos microtúbulos. As membranas duplas são formadas como
parte da autofagia, uma resposta celular à escassez nutricional que
gera autofagossomos para transportar proteínas velhas e organelas
para os lisossomos para degradação. Além de ser um processo celular
normal em condições de escassez nutricional, a autofagia pode ser
ativada pela infecção viral como parte de um mecanismo de defesa
inata; entretanto, este mecanismo anti-viral é sequestrado por
alguns vírus de RNA de fita positiva quando os autofagossomos são
usados para facilitar a montagem de proteínas replicases. NSP6 gera
autofagossomos do RE, mas limita o diâmetro do autofagossomo e a
expansão, o que inibe a capacidade do autofagossomo de transportar
componentes virais aos lisossomos para degradação.
NSP7 e NSP8 – Uma primase
NSP7, NSP8, NSP9 e NSP10 são constituintes do complexo de replicação
do RNA dos coronavírus.
Coronavírus codificam para
duas atividades de RNA polimerase RNA-dependentes. Uma é dependente
de primer e está associada com NSP12 (ver abaixo). A outra está
associada com NSP8, uma proteína de 22kD que é própria do
coronavírus e é capaz de promover a iniciação de novo da
síntese de RNA com baixa fidelidade a partir de moldes de fita
simples de RNA. Por esta razão tem sido proposto que a NSP8 opere
como uma primase, ou seja, ela faz primers de oligonucleotídios que
podem ser usados pela NSP12, que é a principal RNA polimerase
RNA-dependente. NSP7 e NSP8 formam um supercomplexo, uma estrutura
cilíndrica construída com oito cópias de NSP8 fortemente seguradas
juntas por oito cópias de NSP7.
NSP9 – Uma proteína de função desconhecida
NSP9 é uma proteína dimérica
que se liga a RNA de fita simples.
NSP10 – Uma proteína do citoesqueleto
NSP10 é uma proteína do
citoesqueleto com dois zinc-fingers. Ela interage com NSP14 e NSP16
estimulando suas atividades exoribonucleases 3'-5' (NSP14) e
O-metiltransferase 2' (NSP16). NSP10 é requerida pela NSP16 como um
fator estimulatório para executar as últimas atividades de
metiltransferase e pode estabilizar o bolso S-adenosyl
methionine-binding e extender o substrato ranhura de ligação ao RNA
da NSP16.
NSP11
– Uma proteína de função desconhecida
A função desta proteína é
desconhecida, visto que uma deleção no sítio NSP10-NSP11/12 aboliu o
processamento mediado pela protease NSP5, mas permitiu a produção de
partículas virais infecciosas, sugerindo que a clivagem no sítio
NSP10-NSP11/12 não é necessária para a replicação viral em células
cultivadas.
NSP12 – Uma RNA polimerase
Esta proteína - a principal
RNA polimerase - é montada junto com NSP7 e NSP8 em um complexo de
síntese de RNA multi-subunidade que promove a replicação e
transcrição do genoma viral.NSP12 tem uma extensão N-terminal típica
que tem sido proposta como contendo uma atividade
nucleotidiltransferase, enquanto que a replicação do genoma de RNA
viral é catalizado por um domínio polimerase na região C-terminal.
NSP13 – Uma helicase
NSP13 é uma helicase de RNA
e a 5′
trifosfatase que interage com a RNA polimerase NSP12. Uma helicase é
uma enzima que cataliza o desenrolamento de uma duplex de
oligonucleotídios a fitas simples de maneira trifosfato-dependente.
NSP12 aumenta a atividade helicásica da NSP13, mas como a NSP12
aumenta a atividade helicásica é desconhecido.
NSP14 – Uma exonuclease e metiltransferase
A
maioria das RNA polimerases não possuem uma atividade de “revisão de
leitura” e como resultado o tamanho do RNA de vírus é normalmente
limitado a cerca de 10kb (ver abaixo). Entretanto, genomas de
coronavírus são os maiores entre os vírus de RNA com um tamanho de cerca
de 30kb. Devido à taxa de erro das RNA polimerases devidos à
tautomerização de bases, coronavírus aparentemente teria algum tipo de
revisão de leitura.
NSP14, que forma um complexo com NSP10, é uma exoribonuclease e sua
inativação leva ao decréscimo de fidelidade de replicação de 15 vezes.
Ela hidrolisa o RNA de fita dupla na direção 3' a 5' e também um
nucleotídio mal pareado no terminal 3' mimetizando um produto de
replicação errôneo. A atividade exonucleásica está também envolvida na
síntese do conjunto de RNAs genômicos que codificam para proteínas
estruturais. Além dessa atividade nucleásica, a NSP14 também tem uma
atividade metiltransferásica (guanina-N7) envolvida no capeamento 5’ dos
RNAm e do RNA genômico.
NSP15 – Uma endonuclease
NSP15 é uma endonuclease hexamérica que cliva prefencialmente em
uridinas. Ela se associa com a primase (NSP9) e a RNA polimerase
(NSP12). Mutações no sítio catalítico reduz o acúmulo sub-genômico de
RNA e atenuaram profundamente a proliferação viral. Coronavírus são
capazes de evitar a detecção por sensores imunes inatos do hospedeiro
que reconhecem RNAs de fita dupla. NSP15 é requerido para a evasão
desses sensores dsRNA.
NSP16 - A methyl transferase
NSP16 em complexo com NSP10 tem uma atividade RNA ribose 2’-O-metilação.
Para mimetizar a estrutura do RNAm celular, muitos vírus modificam o
terminal 5' de seus RNAs. O 5’ cap é importante para a estabilidade do
RNA, para a tradução a proteínas e também para o escape imune do vírus.
Em adição à metiltransferase NSP14 S-adenosyl-L-methionine-dependent
(guanine-N7) metiltransferase, coronavírus têm outra, a NSP16 que é uma
metiltransferase S-adenosyl-L-methionine (SAM)-dependente ribose
2’O-metiltransferase.
|
|
 Figura 14
Figura 14
Os domínios da proteína (S) da espícula
 Figura 15
Figura 15
A associação das proteínas S, M e E com a membrana viral.
|
As
Proteínas Estruturais
Estas são as proteínas que compõem a partícula
viral madura:
Proteína da Espícula
(S)
A proteína da espícula (S) se projeta
da membrana lipídica viral e é um homotrímero de proteínas S
responsáveis pelo acoplamento dos receptores do hospedeiro e para a
fusão do envelope viral com a membrana da célula hospedeira. Cada
monômero tem uma massa molecular de 175kD e é glicosilada no
terminal N. No SARS-CoV-1 e -2, o trímero se liga ao receptor de
angiotensina humana 2 (hACE2) na superfície da célula após o que o
vírus entra na célula por endocitose.
Cada monômero da proteína da espícula (S)
consiste em duas subunidades (figura 14) formadas pela clivagem
proteolítica usando uma enzima do complexo de Golgi chamada furina.
Estas são as subunidades S1(subunidade de ligação/acoplamento) e a
subunidade S2 (subnidade fusogênica). S1 se liga primeiro aos
receptores de membrana plasmática da célula e depois se dissocia
deles.
A
proteína S atravessa a membrana viral uma vez com o terminal N
glicosilado no exterior do vírus (figura 15).
Proteína de Membrana (M)
A
proteína M tem três domínios transmembrana (figura 15). Ela promove
a curvatura da membrana e forma uma ligação entre as estruturas
internas do envelope e o nucleocapsídio. Ela tem um domínio
N-terminal pequeno localizado fora da partícula viral (que equivale
ao lado luminal das vesículas de membrana intracelulares) e um
grande domínio C-terminal, compreendendo metade da proteína no
interior da partícula viral (esta é equivalente ao lado
citoplasmático da vesícula). Proteínas M de alguns alfacoronavírus
contém um segmento adicional hidrofóbico que funciona como um
peptídeo sinal. A proteína M é glicosilada no seu domínio N-terminal
extracelular. Ela aparenta ser a catalizadora da montagem do vírus,
visto que ela pode interagir com todas as proteínas estruturais,
inclusive com ela própria. Em alguns coronavírus proteínas M e E
podem formar estruturas do tipo vírus, mas no SARS-CoV-1 a proteína
N é necessária também para formar essas estruturas.
Proteína do Envelope (E)
A
proteína E (figura 15) tem um papel na montagem do vírus e na
liberação, e está envolvida na patogênese viral visto que o vírus é
atenuado in vitro e in vivo quando o gene E é
deletado. A proteína E é uma pequena proteína de membrana
multifuncional integral que forma um canal iônico que é importante
nas interações vírus-hospedeiro. Uma vez que a proteína E é pequena,
a oligomerização é necessária para formar o canal. Não se sabe por
que os coronavírus precisam de um canal iônico, mas vários outros
vírus tem proteína similar. No caso do vírus influenza que também
entra na célula por endocitose, aparentemente na acidificação do
endossomo a proteína do influenza M2 permite a transferência de íons
hidrogênio para a partícula viral para auxiliar no desnudamento do
genoma.
A proteína E tem um domínio atravessador de membrana no terminal N
na superfície externa da partícula viral e uma âncora de ácido
palmítico no lado interior ligado a um resíduo de cisteína (figura
15). Ratos infectados com coronavírus que possuem atividade de canal
iônico rapidamente perderam peso e morreram, enquanto os infectados
com mutantes da proteína E que não tinham atividade de canal iônico
recuperaram da doença e a maioria sobreviveu. A proteína E está
envolvida em vários aspectos do ciclo de vida do vírus, tais como a
montagem, o brotamento, a formação do envelope e a patogênese.
|
| |
Proteína (N) do
Nucleocapsídio
Esta proteína interna tem dois
domínios, cada um pode se ligar ao RNA do vírus por mecanismos
diferentes. Ela se liga à NSP3 para acoplar o RNA genômico ao
complexo de replicação-transcrição, e pacotes dos genomas
encapsidiados às partículas virais. A proteína N se liga ao genoma
viral para formar o core de ribonucleoproteína do vírus e interage
com o domínio carbosi-terminal da proteína M.
Hemaglutinina-esterase (HE)
Um gene do lado 5’ da proteína S
codifica para a quinta proteína estrutural, a hemaglutinina
esterase. Ela está presente em alguns beta-coronavírus incluindo o
vírus da hepatite murina, mas não no SARS-Cov-1 ou -2. HE se liga a
ácidos siálicos nas glicoproteínas de superfície e age como uma
hemaglutinina. Ela contém atividade acetil-transferásica que pode
aumentar a entrada na célula mediada pela proteína S e o vírus se
espalha através da mucosa. Ela aumenta a neurovirulência do vírus da
hepatite murina, mas o gene da HE é perdido em cultura de tecidos
por razões não conhecidas.
HE
forma espículas (menores do que as espículas S) na superfície do
vírus. É um dímero e não aparenta ser essencial para a replicação
nos tipos que a possuem. A atividade esterásica da proteína HE pode
clivar o ácido siálico de uma cadeia de carboidrato, o que pode
auxiliar o vírus a escapar da célula na qual ele se replicou.
Anticorpos contra a proteína HE podem também neutralizar o vírus.
|
| |
Outras fases abertas
de leitura (ORFs)
A inspeção dos genomas do SARS-CoV-1 e
-2 mostra uma variedade de fases abertas de leitura (ORFs)
adicionais. Uma ORF é uma região do genoma que tem a capacidade de
ser traduzida a proteína. Para ser traduzida ela precisa de um códon
de iniciação (normalmente AUG) e de um códon de parada (normalmente
UAA, UAG ou UGA). Há alguma informação sobre as funções das
proteínas codificadas nessas ORFs, mas o papel de outras é até o
presente desconhecido.
ORF3a e 3b
As proteínas 3a e 3b são codificadas
por ORF3a e ORF3b e fazem o segundo maior RNA codificado no genoma
do SARS-CoV. A proteína 3a tem-se mostrado presente na membrana
plasmática em um padrão pontilhado, e também intracelularmente. A
região N-terminal da proteína 3a consiste em três domínios
transmembrana. A proteína forma um homotetrâmero e como todas as
proteínas de canal de íon formam homo- ou hetero-polímeros e
associam com a membrana, tem sido proposto que a proteína 3a possa
formar um canal de íon.
A proteína 3b pode ter um papel na imunomodulação. Além disso, ela
pode agir como um antagonista de interferon.
ORF6
A proteína ORF6 é encontrada associada com as
membranas do retículo endoplasmático e complexo de Golgi em células
infectadas por SARS-CoV-1. Esta proteína é indispensável em
replicação viral in vitro, mas funciona na escapada viral do
sistema imune inato, particularmente na inibição da produção e
sinalização do interferon tipo I.
ORF7a
Esta é a menor proteína estrutural que
não é essencial para a replicação do vírus. Ela pode estar envolvida
na montagem viral e no desacoplamento/escapamento de partículas
virais da superfície da célula. Ela deve fazer isso pela inibição da
glicosilação do antígeno estrômico 2 (BST2, também conhecido como
teterina, do inglês ‘tethering’=prender) da medula óssea que
acredita-se restrinja a liberação do vírus da superfície da célula
infectada ao prender a partícula viral envelopada em brotamento na
membrana plasmática.
ORF7b
Esta é outra proteína estrutural
pequena encontrada no complexo de Golgi. Não é essencial para a
replicação viral in vitro ou in vivo.
Orf9b
Esta proteína interfere com as ubiquinização de proteínas celulares
e, como ocorre com a ORF6, altera a imunidade inata hospedeira.
|
|
|

 Figura
17
Figura
17
Coronavírus dentro de vacúolos ligados à membrana e cisternas do
retículo endoplasmático rugoso. Eletromicrografia de camada fina de
células infectadas.
CDC/C.S.
Goldsmith/T.G. Ksiazek/ S.R. Zaki
|
Ciclo de Vida
O ciclo de vida de um Coronavírus é visto na
Figura 16.

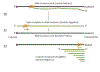

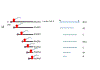
Figura 16. Ciclo de Vida do Coronavírus
Fita de RNA (sentido)
positivo) está em prêto
Fita de RNA
(anti-sentido) negative está em verde
As proteínas são
mostradas em azul e laranja (exceto na última imagem onde S está
em vermelho, E está em púrpura e M está em marrom).
1. A proteína (da
espícula) S do vírus se liga ao receptor (enzima conversora de
angiotensina 2) na superfície de uma célula epitelial.
2. A membrana plasmática
invagina e o vírus entra no endossomo.
3. Íons hidrogênio são
bombeados para os endossomos iniciais para formar um endossomo
tardio.
4. A diminuição do pH no
endossomo muda a conformação da proteína S de forma que a
subunidade S2 (fusógeno) permite que a membrana viral se fusione
com a membrana endossômica. O nucleocapsídio entra no citoplasma
e é desnudado.
5. O genoma viral é de
sentido positivo (mesmo sentido de um RNAm) e tem as
características de um RNAm (Ele é capeado pela 5’ metil
guanosina e tem uma cauda 3’ poli-A). Ribossomos se ligam ao RNA
genômico e traduz os dois terços do genoma da ponta 5’ à
poliproteína p1ab.
6. A poliproteína p1ab
tem uma atividade endoproteásica que corta o protudo primário de
tradução em p1a e p1b.
7. p1a e p1b são
cortadas pela mesma atividade proteásica a 15 ou 16 proteínas
não-estruturais (NSPs).
8. NSPs 7, 8 e 12 se
associam para formar a RNA polimerase viral (replicase).
9. A polimerase se liga
a um sítio de iniciação no terminal 3’ do RNA genômico.
10. O RNA genômico de
sentido positivo é copiado (3’ a 5’) a um RNA genômico de
sentido negativo de comprimento inteiro. Este não é capeado ou
poliadenilado, pois o RNA de sentido negativo não tem capeamento
ou sinal de adenilação.
11. O RNA de sentido
negativo é copiado de 3’a 5’ a RNA genômico de sentido positivo
que é capeado em 5’ e poliadenilado em 3’.
12. O RNA genômico é
copiado pela RNA polimerase a um conjunto localizado de RNAs
sub-genômicos de sentido negativo (sgRNAs).
13. Os sgRNAs de sentido
negativo são copiados a sgRNAs de sentido positivo que são
capeados em 5’ e poliadenilados em 3’ e agem como RNAm.
14. Os sgRNAs de sentido
positivo são traduzidos a proteínas que são na maioria proteínas
estruturais do vírus.
15. A proteina N se liga
ao RNA genômico viral de sentido positivo para formar o
nucleocapsídio e à proteína M que é incorporada nas membranas
intracelulares.
16. O nucleocapsídio
brota através das membranas do retículo endoplasmático/complexo
de Golgi para o espaço luminal.
17. O vírus deixa a
célula junto com a via secretória.
Receptores de
SARS-CoV-1, SARS-CoV-2 e outros Coronavírus
As proteínas S e HE se ligam ao ácido siálico que é encontrado nas
superfícies de todas as células; entretanto, todos os coronavírus
têm um tropismo tissular restrito e assim a ligação é provavelmente
mais complicada. Além disso, alguns coronavírus não se ligam a ácido
siálico e alguns, incluindo o SARS CoV-1 e -2, não tem um gene HE.
Ambos SARS-CoV-1 e SARS-CoV-2 usam o mesmo
receptor celular, embora haja alguma sugestão de que eles se ligam a
uma região discretamente diferente do receptor. A proteína trimérica
da espícula se liga à enzima conversora de angiotensina 2 (hACE2)
após o que o vírus entra na célula por endocitose. Tem sido
encontrado que transformando células Hela para que elas expressem
hACE2 as tornam susceptíveis a infecções pelo SARS-CoV-2.
MERS-CoV usa um receptor diferente, ligando ao
antígeno de superfície celular CD26 (dipeptidil peptidase 4).
No caso do vírus da hepatite murina, o receptor é um membro da
superfamília de imunoglobulinas, molécula de adesão relacionada a
antígeno carcinoembriônico (CEACAM), and anticorpos contra esta
kproteína bloqueia o acoplamento do vírus.
Muitos coronavírus alfa utilizam a
aminopeptidase N (APN) como receptor.
Entrada na célula
Alguns coronavírus, tais como o MHV, podem fusionar com a membrana
plasmática e não requerem endocitose, mas agentes lisossomotrópicos
mostram que a entrada do nucleocapsídeo do SARS-CoV-1 e SARS CoV-2
no citoplasma é via endocitose. Uma vez estando no endossomo ácido,
a proteína S é clivada por uma protease catepsina ácido-dependente
ou outra protease. Isso altera a conformação da proteína da espícula
de forma que o fusógeno (S2) pode promover a fusão das membranas
virais e celulares.
Estudos usando cryo-EM da espícula do
SARS-CoV-1 e sua interação com a hACE2 mostrou que no endossomo
tardio há a dissociação de S1 de hACE2 seguido por uma mudança na
conformação de S2 que é necessária para a fusão da membrana ejetando
assim o nucleocapsídio para o citoplasma.
Fosfoinositídeos estão também envolvidos na
endocitose. Fosfatidilinositol-3,5-bifosfato (PI(3,5)P2) é
sintetizado pela fosfatidilinositol-3-fosfato-5-cinase nos
endossomos iniciais e controla a transição do endossomo inicial para
endossomos tardios. Quando as células que expressam o receptor ACE2
foram tratadas com um inibidor desta enzima (apilimod), houve uma
redução significante da entrada do vírus, enquanto não houve efeito
na entrada do pseudovírus VSV-G, que ocorre nos endossomos iniciais.
Dentro da célula
Coronavírus, como muitos outros vírus de RNA de fita positiva,
rearranjam membranas celulares que são então usadas na replicação e
transcrição de genomas virais (figura 17). Especificamente, NSPs de
coronavírus induzem a formação de vesículas de dupla membrana nas
células infectadas.
Em virtude de ser o vírus de fita positiva,
isto é, do mesmo sentido que o RNAm, os genes do lado 5’ do RNA
viral são traduzidos imediatamente a uma grande poliproteína p1ab
que é então clivada por uma atividade proteásica codificada pelo
vírus presente na poliproteína a p1a e p1b. Estas duas poliproteínas
são processadas a 15 ou 16 (dependendo do tipo de vírus) NSPs por
atividades proeásicas virais múltiplas também presentes nas
poliproteínas. Algumas destas NSPs formam o complexo de
replicação-transcrição (RTCs) que sintetiza o RNA viral.
As proteínas RTC são encontradas em associação
com membranas convolutas e vesículas de dupla membranas que,
juntamente com fatores hospedeiros, copia o genoma a um template de
fita negativa do tamanho completo do genoma do qual novos genomas de
fita positiva serão transcritos. Isto é replicação do genoma.
Alternativamente, o genoma é transcrito a RNAs subgenômicos menores
que são então copiados a RNAs sub-genômicos que codificam para
proteínas estruturais (S, N, E etc.) e outras proteínas acessórias.
Os RNAs sub-genômicos codificam proteínas estruturais do CoV e
proteínas acessórias de função desconhecida.
As proteínas da espícula (S), da membrana (M),
do envelope (E) e do nucleocapsídio (N) são as proteínas principais
dos vírus novos. A proteína N se liga a RNA genômico e permite sua
encapsulação em nucleocapsídios. As proteínas S, M e E são proteínas
da membrana integral que são feitas em associação com os ribossomos
associados ao retículo endoplasmático rugoso. A proteína S, pelo
menos, é clivada por protease de sinal da célula hospedeira. O
domínio C-terminal da proteína M do coronavírus MERS contém uma rede
de sinal de localização trans-Golgi e a proteína 7b tem um sinal de
retenção Golgi. As proteínas incorporadas na membrana migram através
do retículo endoplasmático para o complexo de Golgi onde o vírus é
montado na superfície citoplasmática. A proteína M organiza os
componentes da membrana viral e interage com a proteína do
nucleocapsídio (N) para dirigir a montagem do vírus e o brotamento.
Os nucleocapsídios brotam intracelularmente
para o compartimento intermediário do complexo RE-Golgi e viaja para
o exterior da célula pela via secretória. Isto está em contraste com
a maioria dos outros vírus que se ligam à membrana que brotam
através da membrana plasmática.
Revisão de Leitura
A maioria dos vírus de RNA são pequenos com um
tamanho de genoma de 0kb ou menos. Como visto em outra parte deste
livro, sua RNA polimerase não tem um mecanismo que reconhece e
corrije êrros (mutações) que surgem durante a replicação do genoma.
Como resultado, são formadas quasispécies, que levam ao aparecimento
de adaptação e patogênese. Tais mutações são inevitáveis porque as
formas tautoméricas das bases dos nucleotídeos levarão a uma baixa
taxa de êrros de incorporação pela polimerase, por mais correta que
ela seja. Há claramente uma necessidade de um genoma de tamanho
completo que funcione perfeitamente, mas em um genoma de vírus
pequeno (<10kb), a instabilidade genômica (carga mutagênica) levando
a partículas virais não viáveis não é um grande problema.
Entretanto, quanto maior for o genoma, maior a probabilidade de
surgirem mutações deletérias.
Não se sabe como vírus com grande genoma de
RNA como os coronavírus (mais de 32 kb) lidam com a potencial
instabilidade genômica, mas algum tipo de revisão de leitura como as
que existem nas DNA polimerases, é decerto necessária. Coronavírus
têm várias NSPs que estão envolvidas na síntese de RNA e na formação
de RTCs que são típicas deste vírus. Essas enzimas incluem uma 3′-5′
exoribonuclease, NSP14. A inativação da exoribonuclease do
coronavírus resulta em uma diminuição da fidelidade genômica. Dessa
forma NSP14 pode aumentar a acurácia na síntese de RNA pela correção
de erros de incorporação de nucleotídeos feitos pela RNA polimerase
RNA-dependente do vírus.
|
|
|
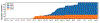 Figura 18
Figura 18
Um gráfico de contagens cumulativas de sequências D614 e G614 por dia no
distrito de Snohomish. Cor laranja representa a forma original, azul a
forma com a mutação G614. 2020. Distrito de King, Estado de Washington,
EUA
Rastreando mutações
da espícula do SARS-CoV-2 (lanl.gov)
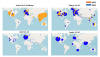 Figura 19
Figura 19
Mapas mostrando a frequência relativa de amostragem de D614 e G614 em
diferentes janelas de tempo. O tamanho dos círculos indica a amostragem
relativa em um dado país dentro de cada um dos quatro mapas. A proporção
do D614 do vírus original de Wuhan é mostrada em laranja e a proporção
do D614 mutante é mostrada em azul. Em cima à esquerda, a distribuição
antes de 11 de março/2020; em cima à direite, a distribuição em 11 de
Março/2020; em baixo à esquerda a distribuição em 11 de Abril/2020; em
baixo à direita a distribuição em 11 de junho;2020.
Rastreando mutações da espícula do SARS-CoV-2 (lanl.gov)
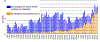 Figura 20
Figura 20
Percentagem de casos positivos para Covid-19 conforme detectado em um
laboratório no sul da Inglaterra entre o início de outubro e metade de
Dezembro/2020. As barras azuis mostram os testes positivos em percentual
de todos os testes realizados. Estes aumentaram de 7% para cerca de 20%
na metade de Dezembro. As barras alaranjadas mostram a proporção de
testes que detactaram a variante B.1.1.7. A variante esteve pouco
presente no início de outubro, enquanto que na metade de Dezembro quase
todos os testes revelaram a variante.
 Figura 21
Figura 21
Os domínios da proteína S do SARS CoV-2 mostrando os sítios de clivagem
pela Furina.
 Figura 22
Figura 22
As mutações importantes na variante B.1.1.7 encontradas originalmente no
sudeste da Inglaterra. Três são deleções e 14 são mutações de ponto.
Elas fazem um cluster nas fases de leitura aberta 1a, S (proteína da
espícula), 8 e N.
.
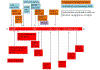 Figura 23
Figura 23
A distribuição de mutações importantes no gene S da variante B.1.1.7 é
vista em vermelho na parte inferior da figura. As mutações na variante
501Y.V2 do Sul da África como detectadas até 15 de outubro/2020 são
mostradas em cima, em laranja. Ao final de novembro a variante adquiriu
mutações adicionais mostradas em azul. Todas estas variantes têm a
mutação D614G. Aminoácidos sombreados estão no domínio de ligação ao
receptor.
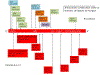 Figura 23a
Figura 23a
A distribuição de mutações importantes no gene S da variante P.1 (em
cima) e da variante B.1.1.7 (em baixo)
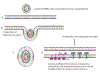
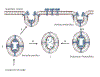
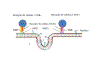 Figura 24 Figura 24
Apresentação da proteína S do SARS CoV2 ao Sistema imune por uma vacina
de RNAm.
O RNAm capeado e poliadenilado é encapsulado no interior de uma
nanopartícula lipídica (A) que é capturada por um endossomo (B e C). O
RNAm é liberado no citoplasma e se associa a ribossomos. A proteína
traduzida contém uma sequência de sinal no terminal N de forma que o
polissomo se associa com o receptor de sinal na superfície
citoplasmática do retículo endoplasmático e transloca para o espaço
intra-retículo endoplasmático. Ela adquire modificações pós-tradução,
incluindo glicosilação, dobra-se da forma normal e trimeriza (D). A
proteína S segue a rota exocítica normal via Complexo de Golgi e passa
pelos exossomos (E). Daí a proteína pode ir para a superfície da célula
(F) e adentrar por endocitose em um endossomo proteolítico (G) ou pode
ir diretamente para um endossomo proteolítico sem secreção (H). A
proteína é degradada por proteases endossomais e os peptídeos
resultantes são ligados a antígenos do complexo maior de
histocompatibilidade (MHC) (I)Ocorre então a ativação de células T CD4+
e CD8+ via apresentação dos peptídeos no MHC de classe II e de classe I,
respectivamente (J).
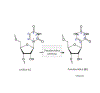 Figura 25
Figura 25
Estrutura da pseudouridina e uridina.
 Figura 26
Figura 26
Transcrição de uma molécula de vacina de RNAm a partir de um construto
de plasmídeo.
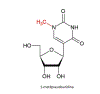 Figura 27
Figura 27
1-methylpseudouridine. Um grupo metílico estra é adicionado
enzimaticamente à base do pseudouracil.
Ver também: VIROLOGY CHAPTER 8
VACCINES |
VARIANTES
DO SARS COV-2
SARS CoV-2 pertence à família dos Coronavírus que tem o maior genoma
entre todos os vírus de RNA. Apesar de terem alguma capacidade de
revisão de leitura, esses vírus, assim como todos os vírus de RNA, são
sujeitos a rápidas mutações quando eles replicam. A maioria das mutações
são deletérias para o vírus ou são silenciosas (isto é, não têm efeito).
Esta última não altera a sequência de amonoácidos (só alteram a
sequência de nucleotídios) ou são mutações conservativas nas quais as
propriedades das cadeias laterais dos aminoácidos são similares (ex. uma
mudança de alanina para leucina ou ácido aspártico para ácido
glutâmico). Entretanto, algumas mutações dão ao vírus uma vantagem
seletiva; eles irão proliferar mais rapidamente e irão se tornar
dominantes na população rapidamente.
Mutação D614G
O vírus
original de Wuhan tem um ácido aspártico na posição 614 na região de
ligação ao receptor da subunidade S1 da proteína da espícula (614D).
Este mutou para uma forma mais infecciosa com glicina na mesma
posição. O vírus mutante (conhecido como D614G, ou simplesmente
G614) aumentou de frequência relativa à 614D de uma forma
consistente com uma vantagem seletiva. Este tipo substituição de
aminoácidos por mutação se tornou cada vez mais comum à medida que o
vírus SARS-CoV-2 se espalha pelo mundo. De fato, os vírus originais
G614 SARS-CoV-2 diferiram da forma original de Wuhan por 4 mutações
e quase sempre G614 é encontrada ligada a outras 3 mutações.
A forma de vírus D614 de Wuhan se espalhou rapidamente pelo globo no
início de 2020, mas quando D614 e G614 co-circularam a forma G614
normalmente mostrava um crescimento rápido na frequência relativa e
veio a dominar a população de vírus. D614G é agora claramente a
forma dominante do vírus globalmente e a transição levou cerca de
4-6 semanas (figura 18 e 19).
Uma questão surgiu sobre porque G614 pareceu sobrepor D614. Isto
pode ser devido ao conhecido efeito do fundador em que a forma
mutante surgindo em um super disseminador faz com que haja mais
desses vírus disponível para infectar outras pessoas.
Alternativamente, a forma mutante poderia ser simplesmente mais
infecciosa (isto é, transmissível) do que a forma D614. Esta última
parece ser o caso, visto que a frequência de G614 aumentou em todos
os lugares em Março/2020, incluindo em muitas áreas onde G614
apareceu em epidemias locais de G614 bem estabelecidas. Uma
investigação profunda da transmissibilidade no Reino Unido encontrou
que G614 aumenta em frequência relativa à D614 de maneira
consistente com uma vantagem seletiva no vírus.
Variante do UK B.1.1.7 Dezembro 2020
Variantes surgem a toda hora com as mutações do
vírus, mas em Dezembro/2020 uma variante chamada B.1.1.7 foi
identificada que apareceu mais frequentemente em amostras no sul da
Inglaterra, embora esta variante tenha estado de fato circulando por
alguns meses. Quando comparada com ovírus de Wuhan, esta variante
contém 23 mutações. Algumas são silenciosas, mas algumas podem
afetar a interação da proteína S do vírus com o receptor ACE2 da
célula. Esta variante deslocou outras variantes à medida que ela se
espalha pelo sudeste da Inglaterra, sugerindo que é mais facilmente
transmissível (mais do que D614G que já era mais transmissível do
que o vírus original de Wuhan) embora outras explicações do
deslocamento sejam possíveis. É estimado que B.1.1.7 tem uma taxa de
transmissão aumentada de 50 a 70 porcento comparada com outras
variantes. Embora a variante se espalhe mais rapidamente, não há
evidência de que ela cause doença mais severa ou de que ela não seja
suscetível a vacinas que tenham como alvo originalmente a D617G.
A proteína S consiste em 1273 aminoácidos. No corpúsculo de Golgi
ela é clivada por uma protease chamada Furina às subunidades S1 e
S2. Há dois sítios de clivagem de Furina e uma parte da proteína é
perdida. A subunidade S1 contém um peptídio sinal no terminal N e o
domínio de ligação ao receptor. A subunidade S2 contém a sequência
de fusão que permite o envelope viral se fusionar com a membrana
celular, o domínio transmembrana e o domínio citoplasmático (figura
21).
B.1.1.7 tem 23 mutações não sinônimas (mutações que fazem com que um
aminoácido mude ou pare a síntese protêica. A Figura 22 mostra as
mutações de importância na variante. Oito dessas mutações estão no
gene da proteína S incluindo duas pequenas deleções. Duas são no
domínio de ligação ao receptor da proteína S; estas são N501Y que
causa uma mudança de Asn a Tir (ambos aminoácidos polares neutros,
embora a cadeia lateral da Tir seja maior) e A570D em que a mudança
é de Ala para Asp. Esta é uma mudança não conservativa na qual um
aminoácido polar neutro é substituído por aminoácido polar. Esta
mutação pode ser a base de maior transmissibilidade da variante pois
ela pode alterar interação proteína S - receptor. Três mutações são
em uma ORF (fase de leitura aberta) 8, uma das quais é uma mutação
de parada levando a uma proteína truncada inativa. Entretanto, a
deleção ORF8 tem apenas um pequeno efeito na replicação do vírus,
comparado a vírus sem a deleção.
No final
de dezembro/2020 esta variante foi identificada em vários países a
nos Estados Unidos. Nenhuma das duas primeiras variantes positivas
americanas viajaram internacionalmente nas semanas recentes.
Variante Sul-Africana 501Y.V2Ao mesmo tempo em que B.1.1.7 estava emergindo
como dominante, a variante mais transmissível no sudeste da
Inglaterra, outra mais transmissível variante estava surgindo no sul
da África. Esta é conhecida como 501Y.V2 e tem oito mutações
definidoras de linhagens na proteína da espícula, incluindo três em
aminoácidos importantes no domínio de ligação ao receptor que pode
ter importância funcional. Essas mutações são mostradas na figura 23
onde são comparadas com as de B.1.1.7. As três mutações importantes
em 501Y.V2 são K417N (Lis para Asn, básico para neutro polar), E484K
(Glu to Lis, uma mudança de um ácido para um básico) e N501Y (Asn
para Tir, ambos aminoácidos neutros polares). A mutação N501Y é
também vista no gene da proteína S de B.1.1.7 e é parte do loop de
ligação na região de contato com a ACE2 humana, onde forma uma ponte
de hidrogênio com a tirosina 41 do ACE2. Ela também interage com a
lisina 353 na região ligadora ao vírus de ACE2 e pode aumentar a
afinidade de ligação do SARS-CoV-2 pelo ACE2 humano. Há alguma
evidência de que a mutação E484K também possa modestamente aumentar
a afinidade de ligação do receptor, mas a mutação K417N tem pouco
efeito na afinidade de ligação da ACE2.
A variante, como seria esperado, acumulou mais
mutações com o tempo. Em 15 de Outubro a variante sulafricana tinha,
além da D614G, cinco outras mutações não sinônimas na proteína da
espícula: D80A, D215G, E484K, N501Y e A701V. Três mutações de
espícula adicionais emergiram ao final de novembro: L18F, R246I e
K417N (figura 23).
Alterações na variante B.1.1.7 - janeiro 2021
A original B.1.1.7
não contém a mutação de escape (E484K) que torna a variante
sulafricana mais resistente a vacinas. E484K faz com que seja mais
difícil para os anticorpos se acoplarem ao vírus e impedí-los de
entrarem nas células. No final de janeiro de 2021 algumas variantes
B.1.1.7 na Grã-Bretanha parecem haver adquirido a mutação E484K.
Variante P.1 (B1.1.248)
Em Abril/2020 Manaus, uma cidade na Amazônia
brasileira, sofreu uma severa primeira onda de infecções pelo
SARS-CoV-2, mas a população resistiu a lockdowns e distanciamento
social não foi seguido. Como resultado, foram tantas as pessoas
sendo infectadas (76% da população) que se pensou que a cidade teria
chegado a uma imunidade de rebanho, visto que eles pensavam ter
proteção contra o vírus. Como resultado da imunidade de rebanho
chegando a três quartos da população infectada na onda inicial do
vírus, foi esperado que não haveria muita disseminação do vírus em
uma segunda onda na qual o número Rt seria menor que 1. Entretanto,
em Janeiro de 2021, Manaus sofreu uma segunda onda de infecção da
COVID-19 que congestionou seus hospitais deixando os suprimentos de
oxigênio exauridos e dezenas de pessoas a morrerem em suas casas e
em hospitais de cuidados intensivos. O sequenciamento mostrou que
uma nova variante de SARS-CoV-2, conhecida como P.1, foi responsável
por cerca de metade das novas infecções. P.1 foi também encontrada
em poucos casos no Japão, entre pessoas que haviam viajado
recentemente de Manaus. Assim como outras variantes, tais como as
inicialmente identificadas em UK e na África do Sil, P.1 aparenta
ser mais transmissível do que a do vírus original D614G que se
espalhou pelo mundo, causando preocupações sobre um risco maior de
disseminação. O vírus tem 17 mudanças de aminoácidos típicas, 3
deleções e 4 mutações sinônimas, e mais uma inserção de 4
nucleotídios comparado com os vírus mais estreitamente relacionados.
Como em outras variantes, mutações no sítio de ligação da proteína S
são as que mais preocupam. São elas a K417N, E484K e N501Y (figura
23a). No caso da lisina na posição 417, há uma mudança para Asn na
variante sulafricana e para Thr na variante P.1. N501Y é encontrada
na variante de UK e sulafricana e mudanças em 417 e 484 estão também
na variante sulafricana. No final de janeiro/2021 a variante P.1 se
espalhou pelo Japão, Alemanha e Estados Unidos.
Então, por que uma
segunda onda de infecções? Seria porque P.1 não é reconhecido por
anticorpos em pessoas que foram infectadas durante a primeira onda
de infecção? Poderia ser que as pessoas presumivelmente imunes por
causa da primeira infecção foram reinfectadas sugerindo que a
imunidade que eles desenvolveram durante a primeira onda não foi
capaz de suprimir a nova variante. Isso é muito preocupante para a
eficácia de vacinas. Entretanto, nós não sabemos (Janeiro 2021) se
as pessoas estão sendo reinfectadas ou se o vírus mais altamente
transmissível está se espalhando para o quarto restante da
população, visto que no final de Janeiro de 2021 houve apenas um
caso confirmado de reinfecção; poderia ser que a transmissibilidade
aumentada tenha elevado o Rt e portanto o patamar do início da
imunidade de rebanho.
Além de fazer o vírus mais transmissível,
aparentemente as mutações P.1 diminuem a capacidade do sistema imune
de reconhecer e neutralizar o vírus. Isso também parece ser o caso
com a variante sulafricana que é tão similar a P.1 em três sítios
importantes na proteína S. Estudos sobre se a variante sulafricana
poderia ser neutralizada com anticorpos de pacientes infectados com
versões antigas do SARS-CoV-2 mostraram que em certa da metade dos
casos a nova variante foi resistente à neutralização pelo plasma do
soro; entretanto, deve ser observado que enquanto P.1, como as
outras variantes, é mais altamente transmissível, não há evidência
atualmente de que ela cause doença mais severa.
Mutações em P.1 (figura 23a)
N501
A mutação em N501 permite a proteína S do vírus se ligar mais
facilmente ao receptor ACE2 na superfície da célula. Isso torna o
vírus mais infeccioso (até 70% mais infeccioso em alguns estudos).
E484
A mutação em P.1 no aminoácido 484 (Glu para Lis) é mais
preocupante. Ela tem sido referida como mutação de escape e está
também na variante sulafricana, mas não na do UK. Ela parece
permitir que o vírus escape pelo menos parcialmente dos anticorpos
gerados em uma infecção prévia não-P.1 e também possivelmente os
anticorpos nos cocktails terapêuticos monoclonais feitos por
companhias como a Regeneron. É de grande preocupação que esta
mutação possa permitir que o vírus escape aos anticorpos gerados
pelas vacinas atuais, que iria necessitar a alteração das sequências
de DNA usadas para gerar tais vacinas. É provável que a vacina
trabalhe contra as novas variantes, evitando a doença pelo COVID-19
séria, que será substituída por sintomas mais amenos.
K417
Ambas as variantes P.1 e a sulafricana têm uma mutação no aminoácido
417, embora os aminoácidos alterados difiram nas duas variantes (Thr
na variante P.1 e Asn na sulafricana). O fato de as mutações ao que
sabemos devem ter surgido independentemente sugere-se que estas
confiram alguma vantagem para o vírus, mas sua importância é
desconhecida.
Variante P.2
Em 12 de
Janeiro/2021, pesquisadores no Brasil relataram a detecção de uma
variante da linhagem P.1 que, como na variante P.1 tem a mutação
E484K. Ela provavelmente evoluiu intependentemente da variante P.1.
São essas variantes neutralizadas pelas vacinas atuais?
A resposta parece ser sim, embora a variante
sulafricana possa ser menos suscetível a ambos os antibióticos
produzidos em uma infecção natural e pelas primeiras vacinas, mas
pelo menos elas previnem doença séria.
Co-infecções
Dois
casos de COVID-19 foram descobertos no Brasil em pessoas com seus 30
anos de idade que foram infectadas pelas duas variantes P.2 e uma
linhagem diferente que circula no Brasil. É possível que essas
co-infecções possam levar à criação de variantes híbridas
adicionais.
VACINAS
CONTRA A COVID-19
Até a pandemia pelo Covid, todas as vacinas bem
sucedidas foram baseadas em vírus atenuados, partículas virais mortas ou
proteínas purificadas (vacinas de subunidades). Estas apresentam
proteínas virais com ou sem o contexto viral ao sistema imune. Elas
requerem muito desenvolvimento, leva tempo para produzir em grandes
quantidades, requerem uma purificação substancial e normalmente não
apresentam o antígeno ao sistema imune da mesma maneira que uma infecção
natural, que resulta em uma célula infectada por vírus. Até
dezembro/2020, nenhuma vacina tinha sido aprovada para uso humano que
fosse baseada na injeção de ácidos nuclêicos, embora essas vacinas sejam
mais fáceis de serem produzidas em grandes quantidades e possam ser
rapidamente acompanhadas para mudanças no vírus circulante usando
técnicas de biologia molecular.
Vacinas baseadas em ácidos nuclêicos pode ser de
DNA ou de RNA. Vacinas de DNA consistem no gene apropriado inserido em
um vetor viral que pode ser capturado pela célula, transcrito o RNAm e
traduzido a proteína. Vacinas de RNA omitem a primeira etapa e inserem
diretamente um RNAm traduzível no citoplasma da célula. Ambos os tipos
de vacina fazem com que a célula produza e processe proteínas virais ou
proteína da mesma forma que ocorre em uma infecção natural. A proteína
de superfície codificada pelo ácido nuclêico passa através das vias de
exportação das células adquirindo as modificações pós-tradução que
também ocorre na infecção natural. O antígeno pode ser também passado
através das vias proteossomais ou proteolíticas resultando em peptídios
que podem ser apresentados na superfície celular em
associação com antígenos de histocompatibilidade de classe I e classe II
e assim mediar uma forte resposta imune mediada por células e também uma
resposta mediada por anticorpos.
VACINAS
DE RNAm
As primeiras duas vacinas aprovadas no fim de 2020
são baseadas em um protocolo no qual o RNAm codificando para o antígeno
de interesse envolvido por um carreador lipídico (nanopartícula
lipídica) é injetado na pessoa que se vacina. O lipídio protege o RNAm
contra ribonucleases e facilita sua entrada nas células. O RNAm é
traduzido a proteína, processado e apresentado ao sistema imune da
maneira usual. A proteína de interesse é normalmente a que se liga ao
receptor da célula e anticorpos contra esta proteína - que bloqueiam a
interação vírus-receptor da célula - irá impedir a infecção e são
chamados de anticorpos neutralizantes. No caso de vacinas contra o
SARS-CoV-2, este é o antígeno S que liga ao receptor ACE2 humano.
Um grande problema com as vacinas de RNAm é sua
estabilidade em trânsito do local de fabricação fora da célula até o
local da infeção e dentro da célula. O DNA, inerentemente estável dentro
da célula, visto que ele passa o código genético de uma célula para
outra indefinidamente. Ao contrário, RNAm têm uma vida muito curta
comparada com a do DNA. A quantidade de um RNAm depende do equilíbrio
entre a taxa de síntese e a taxa de degradação. Muitas proteínas são
requeridas somente por um tempo muito curto, e se seus RNAm fossem muito
estáveis o nível de proteínas não poderia ser controlado. Porisso,
embora todos os RNAm tenham vida curta, muitos são degradados muito
rapidamente após a tradução, facilitando respostass rápidas às condições
na célula. Os RNAm são degradados por ribonucleases (RNAses). Diferentes
RNAm têm diferentes graus de estabilidade, resultando de suas estruturas
secundárias e da natureza dos terminais da molécula. Estes são
conhecidos como elementos cis. Além disso, sua estabilidade é também
regulada pelos fatores de ligação ao RNA ou elementos trans. Elementos
cis incluem a cauda 3’ poli-A e o 5’ metil-guanosina cap. A cauda 3’
poli A é ligada pelas proteínas ligadoras de poli A que estabilizam o
RNA. Essas proteínas requerem um certo comprimento de cauda poli A para
ligar e assim quanto mais longa a cauda poli A mais dessas proteínas
podem ligar ao RNA. O RNAm é degradado do lado 3’ por uma exonuclase e
do lado 5’ pela remoção do 5’ cap e pela ação exonucleásica da 5’-3’
exonuclease. A atividade endonucleásica também degrada RNAm e isso pode
ser regulado por outras proteínas ligadoras a RNA. Sequências ricas em
AU na região 3’ não traduzida (UTR) estão também envolvidas na
estabilidade.
RNAm pode ser também
estabilizado por modificações químicas das bases do ácido nuclêico
propriamente. Tais modificações incluem metil adenosina, a
N-1-metilpseudouridina e pseudouridina (feita da
uridina pela pseudouridina sintetase (figura 25), uma modificação de
base que é comum no RNAt e aumenta a estabilidade. No RNAm estas bases
substituídas aumentam a tradução. Pseudouridina e N-1-metilpseudouridina
reprimem os acionadores de sinalização intracelulares da ativação da
proteína-cinase, que estão envolvidos na estabilidade do RNAm.
Evidentemente, tais modificações não devem alterar a fidelidade da
tradução da mensagem.
Vacinas de RNAm são feitas pela transcrição de um
plasmídio que codifica para uma proteína reconhecida por um anticorpo
neutralizador, no caso de uma vacina contra Covid-19 é a proteína S. O
plasmídio, o qual contém as sequências de promotor apropriadas, é
linearizado e transcrito in vitro usando a RNA polimerase de um
fago T7, T3 ou Sp6. O produto resultante contém uma fase aberta de
leitura que codifica para a proteína S flanqueada por UTRs 5’e 3’, por
uma 5’ metil guanosina cap e uma cauda poli A. Isso é utilizado como
vacina.
A figura 26 mostra uma maneira pela qual isso pode
ser feito em um sistema da AmpTec O gene da proteína S é clonado em um
sítio de inserção em um plasmídio m13 junto com o promotor (A) de T7. Um
primer sentido complementar ao terminal da sequência do M13 (Pri) e um
segundo primer reverso (anti-sentido) complementar ao final do gene S
são usados (B). Este último primer inclui uma sequência poli T,
normalmente em torno de 120 nucleotídios que não hibridiza com nenhuma
sequência de m13. Usando PCR, a estrutura de DNA mostrada em C é
produzida. Este é então usado em transcrição in vitro a partir do
promotor de T7 para formar um RNAm poliadenilado, mostrado em D. A
transcrição in vitro pode acontecer na presença de nucleotídeos
modificados tais como o pseudouracil e/ou N6-metil adenosina, 5-metil
citidina e outros. Estes RNAm modificados são muito mais estáveis do que
os RNAm normais e são altamente traduzíveis conferindo à vacina uma
eficácia muito maior.
A proteína resultante é processada da maneira
normal através da via exocítica com todas as modificações pós-tradução
usuais incluindo glicosilação e transportada para a cuperfície da
célula. Como descrito acima, a proteína pode também ser clivada por
proteases para formar pequenos peptídeos que podem ser apresentados na
superfície da célula ao sistema imune. As células têm mecanismos
anti-virais para detectar e degradar RNAs estranhos e passos têm sido
dados para minimizar isso.
Mesmo com as modificações de nucleotídeos, o RNAm
nu é muito provável que seja rapidamente degradado quando injetado na
pessoa que se vacina. Além disso, o RNAm pode cruzar a membrana celular
para ganhar acesso à maquinária de tradução a proteínas da célula. Ambos
os problemas podem ser resolvidos pela encapsulação do RNAm em um
envelope lipídico (uma nanopartícula lipídica ou lipossomo) que ajuda a
vacina de RNAm a entrar no citodplasma direto do endossomo sem ser
degradado em um lisossomo.
As vacinas
iniciais de RNAm contra Covid da BioNtec e Moderna usam uma tecnologia
similar à descrita acima. Uma modificação que pode ser usada em vacinas
de RNAm futuras é fazer uma vacina de RNAm que contenha não apenas RNAm
para a proteína de escolha (ex. a proteína S do SARS-CoV-2), mas também
um RNAm para uma RNA polimerase (replicase) viral RNA-dependente. Quando
este tipo de vacina de RNAm for injetado em uma pessoa que se vacina, ao
entrar na célula, ele será traduzido a proteína S e a replicase (que
deverá ser codificada no mesmo RNAm ou em um segundo RNAm). A replicase
viral pode reconhecer sinais de replicação incluídos nas vacinas de RNAm
e podem então amplificar o poder da vacina de RNAm, fazendo mais cópias
do RNAm e portanto, mais cópias da proteína. Visto que há agora mais
vacinas de RNAm na célula do que o que era originalmente enviado ao
citoplasma, isto se chama de abordagem RNAm auto-amplificante (SA) (do
inglês self-amplifying).
Vacina
Covid-19 Pfizer-BioNTech: Tozinameran (BNT162B2.
Nome
comercial: Comirnaty)
Tozinameran foi a primeira vacina de RNAm a
ser aprovada. Em estudos clínicos sua eficácia é em torno de 95% 28
dias após a primeira dose e é bem tolerada. Em um dos estudos
iniciais houve 170 casos confirmados de Covid-19dos quais 162 foram
no grupo placebo e 8 no grupo vacina. Ela é ministrada em duas doses
com três semanas de diferença. Não foi avaliada para infecção
assintomática. Ela aparenta ser eficiente contra as variantes
descritas acima. Esta vacina precisa ser armazenada e transportada a
-70 C.
Contém (Programa de
Denominação Comum, OMS):
Uma estrutura modificada
5’-cap1(m7G+m3'-5'-ppp-5'-Am)
Região 5´- não traduzida derivada do RNA da alfa-globina humana com
uma sequência Kozak optimizada. Esta última assegura que a proteína
é corretamente traduzida pelo ribossomo e funciona como sítio de
início de tradução na maioria dos RNAm eucarióticos.
Peptídio sinal da glicoproteína S necessário para dirigir o complexo
proteína/ribossomo nascente até o receptor de sinal na superfície
citoplasmática da membrana do retículo endoplasmático rugoso. Isto
guia a translocação da proteína para a orientação correta no
retículo endoplasmático.
Sequência de códons optimizada codificadora da proteína de tamanho
completo do SARS-CoV2 S que contém duas mutações: K986P e V987P.
Estas alteram o dobramento da proteína S de forma que ela adote uma
conformação pré-fusional antigenicamente optima. Todas as uridinas
são substituídas por resíduos de 1- metil-3’-pseudouridina (Ψ)
(figura 27) que são não obstante, traduzidas eficientemente.
Ao final da sequência codificadora existem dois códons de parada
ΨGA.
A região não traduzida 3´compreende dois elementos de sequência que
conferem estabilidade ao RNA e elevada expressão de proteínas.
Uma cauda poli A com 110 nucleotídeos
consistindo de um trecho de 30 resíduos de adenosina seguido de uma
sequência ligadora de 10 nucleotídeos e outros 70 resíduos de
adenosina.
Além destes a vacina contém lipídios que
formam as sólidas nanopartículas que encapsulam o RNAm (ALC-0315 =
((4-hidroxibutil)
azanediil)bis(hexano-6,1-diil)bis(2-hexildecanoato); ALC-0159 =
2-[(polietilenoglicol)-2000]-N,N-ditetradecilacetamida;
1,2-Distearoil-sn-glicero-3-fosfocolina; e colesterol. Em adição, a
vacina contém água, sucrose, fosfato de sódio dibásico de-hidrato,
fosfato de potássio monobásico, cloreto de potássio e cloreto de
sódio.
Vacina Moderna. RNAm 1273
A vacina Moderna é também de RNAm consistindo
em uma mensagem sintética codificadora da glicoproteína pré-fusão
estabilizada da espícula do vírus SARS-CoV-2 vírus. A estabilização
pré-fusão é conseguida pela substituição de duas prolinas como na
vacina da BioNTech.
Novamente, o RNAm é feito pela transcrição de
um promotor de T7 em uma reação em que UTP foi substituído por
1-metilpseudoUTP. Além do RNAm, a vacina contém lipídios para formar
uma nanopartícula lipídica: (SM-102,
1,2-dimiristoil-rac-glicero3-metoxipolietileno glicol-2000
[PEG2000-DMG], colesterol, e 1,2-distearoil-snglicero-3-fosfocolina)
e, trometamina, cloridrato de tromethamine, ácido acético, acetato
de sódio, sucrose e água.
A eficácia do 1273m é em torno de 94.1%,
similar ao da vacina da BioNTec. Em um estudo inicial, houve 196
casos confirmados de Covid-19 dos quais 185 foram no grupo placebo e
11 no grupo vacina.
Els tem como vantagem sobre a anterior a
formulação da nanopartícula lipídica, que permite que ela seja
armazenada e transportada a 2-8C, e não -70C como é o caso da vacina
BioNTec. É administrada em duas doses, três semanas entre estas.
VACINAS DE DNA BASEADAS EM
ADENOVÍRUS
Vacina: AZD1222, CHADOX1
NCOV-19 – AstraZenica/Universidade de Oxford
A vacina ChAdOx1 nCoV-19 (AZD1222) consiste no vetor de
adenovírus símio deficiente em replicação ChAdOx1 contendo o gene
completo da glicoproteína S do SARS-CoV-2, com uma sequência lider
ativadora de plasminogênio tissular. ChAdOx1 nCoV-19 expressa uma
sequência codificadora com códon optimizado para a proteína S. Um
adenovírus símio e não o humano é usado porque o uso de adenovírus
humano é limitado pela imunidade pré-existente ao vírus na população
humana, que reduz significantemente a imunnogenicidade de vacinas
baseadas no vírus humano. Isto não é problema com o vírus símio,
embora adenorírus símios são estreitamente relacionados aos
adenovírus humanos, as regiões hipervariáveis dos imunógeno
principais são significantemente diferentes das do vírus humano,
evitando assim imunidade pré-existente. O vetor adenovírus símio
não tem a região E1 codificadora para as proteínas virais
transativadoras, que são essenciais para a replicação do vírus e a
região E3 codificadoras de proteínas imunomodulatórias. Deleção
desta última permite a incorporação de sequências genéticas maiores
no vetor viral.
A vacina de adenovírus é capturada por células e o material genético
é transcrito no núcleo para produzir o RNAm que é traduzido à
proteína S. A eficácia é de até 90%, dependendo da dosagem. Maior
eficácia foi encontrada em um subgrupo no qual a primeira das duas
doses foi dada pela metade. A eficácia média foi de 70.4%.
AD5-NCOV, Convidicea (Cansino Biologics, China)
Esta é outra vacina baseada em adenovírus. É
baseada no adenovírus humano recombinante deficiente em replicação
vetor tipo 5 para induzir uma resposta imune. Novamente, o vírus foi
tornado deficiente em replicação pela deleção dos genes E1 e E3. Ele
codifica para um gene de tamanho completo da proteína S baseado na
sequência do vírus Wuhan-Hu-1 com gene do peptídeo sinal ativador de
plasminogênio tissular.
GAM-COVID-VAC, Sputnick V (Gamaleya Research Instituto de
Epidemiologia e Microbiologia, Russia)
Gam-COVID-Vac é uma vacina baseada em dois
vetores modificados de adenovírus humano contendo o gene que
codifica para a proteína S do SARS-CoV-2. A primeira inoculação usa
adenovírus 26 (Ad26) como o vetor para o gene da proteína S,
enquanto o segundo usa adenovírus 5 (Ad5). Esta vacina demonstrou em
janeiro de 2021 ter uma eficácia de 91,6% contra Covid-19
sintomática.
AD26.COV2.S, JNJ-78436735 (Janssen/Johnson e Johnson, Estados Unidos
e Bélgica)
Esta vacina é baseada também em um vetor adenovírus modificado
recombinante. Como a vacina Sputnick, usa Ad26 humano expressando a
proteína S, neste caso em uma única inoculação. Ela produz
anticorpos neutralizante poderosos e resposta mediada por células.
Usa a tecnologia AdVac, que aumenta a estabilidade de forma
que a vacina pode ser armazenada na temperatura de refrigerador por
pelo menos três meses.
VACINAS
DE SUBUNIDADES
NVX-CoV2373, Novavax
A vacina Novavax (NVX-CoV2373) é baseada em tecnologia mais antiga
usando a proteína S do SARS-CoV-2 S com um adjuvante Matriz M. Em
Estudos Clínicos ela produziu altos níveis de anticorpos anti-
proteína S e tem sido adquirida por vários governos como parte de
suas estratégias anti-Covid-19. O gene da proteína S é inserido em
um baculovírus. A família Baculiviridae é uma família de vírus de
DNA circular dupla fita (80-180 pares de bases) que infectam insetos
e artrópodos. O baculovírus modificado é então usado para infectar
células de insetos (comumente células Sf9 isoladas de Spodoptera
frugiperda, (a lagarta-do-cartucho) que faz a proteína S. Esta forma
trímeros nativos na superfície da célula infectada. Estas proteínas
são extraídas e associadas com nanopartículas lipídicas e assim a
proteína S é apresentada ao sistema imune da maneira similar à que
ocorre na superfície de uma célula infectada. Um adjuvante extraído
da Quillaja saponaria Molina 'Soap bark tree' que como o nome em
inglês indica (soap = sabão) pode ser usada como sabão. No caso de
vacinas, ele estimula a atração de células imunes para o local da
injeção onte elas irão responder mais eficientemente. As
propriedades adjuvantes vêm das saponinas (glicosídeos triterpenos).
As nanopartículas que contêm a proteína S são capturadas pelas
células apresentadoras de antígenos, clivadas em peptídeos e
apresentadas na superfície como antígenos em associação com o MHC a
células T e B.
Estudos de fase 3 têm mostrado que esta vacina tem 89%
de eficácia contra o Covid-19 e aparentemente proporciona forte
imunidade contra as variantes do Reino Unido e do sul da África.
VACINA DE PARTICULAS VIRAIS INATIVADAS
Vacina valneva (VLA 2001)
Esta usa uma tecnologia muito mais estabelecida similar à usada
na vacina Salk da pólio, que é o uso de partículas virais totais
inativadas. O vírus é crescido em células (Vero) de rins do
macaco verde africano, purificados e inativados com um agente
tal como a formalina. A vacina também contém adjuvantes alum e
CpG 1018. CpG 1018 é um receptor agonista tipo-toll 9 (TLR9).
OUTRAS VACINAS
ITMV-083, Instituto Pasteur
Esta é uma vacina de vírus atenuado
usando o vírus da vacina do sarampo com um vetor que expressa a
proteína antigênica S do vírus SARS-CoV-2 vírus. Devido à baixa
eficácia, o desenvolvimento desta vacina foi abandonado.
Há várias outras
vacinas contra o SARS-CoV-2 em estudos de fase I e II incluindo
tecnologias antigas, tais como partículas de vírus totais inativados
por formalina (Sinovac e Sinopharm).
POR QUE
PRECISAMOS DE DUAS INOCULAÇÕES?
A maioria das vacinas que são desenvolvidas contra
o SARS-CoV-2 requerem duas inoculações. Isso é por causa da maneira que
o sistema imune responde a patógenos estranhos, tais como um vírus
infectante.
Inicialmente, é importante suprimir a infecção
impedindo que o patógeno entre nas células e se replique. A infecção por
um vírus que se liga ao seu receptor na superfície da célula (ACE2 no
caso do SARS-CoV-2) dispara uma resposta inicial na qual plasmócitos
(linfócitos B|) produzem anticorpos neutralizantes que se ligam à
superfície do organismo invasor como no caso do SARS-CoV-2, bloqueando a
ligação da proteína S do vírus a ACE2. A resposta por anticorpos,
entretanto, declina rapidamente, mas algumas das células B se
diferenciam em células B de memória que sobrevivem por muito, muiito
tempo e se re-alocam na periferia do corpo. Aí elas irão mais
provavelmente encontrar mais antígenos durante uma segunda exposição.
Quando isso acontece, elas proliferam e diferenciam em mais células B
que irão responder ao antígeno produzindo mais anticorpos. Células B de
memória podem sobreviver por muitos anos de forma que elas são capazes
de responder a exposições múltiplas do mesmo antígeno.
Durante a primeira fase da resposta imune, as
células imunes também secretam citocinas, que recrutam outras células
imunes para o local da infecção, entre elas estão as células auxiliares
CD4-positivas e células T citotóxicas (células T assassinas) que
reconhecem e matam células infectadas por vírus. E como as células B,
células T se diferenciam em células de memória que podem reativar e
proliferar em resposta a nova exposição ao antígeno original. Estas
células T de memória podem também permanecer no corpo por muitos anos (e
talvez pela vida inteira).
Visto que
somente um número pequeno de células T de memória são feitas como
resultado da exposição inicial, uma segunda exposição ao antígeno
(infecção ou inoculação) é necessária para amplificar o seu nível.
Assim, com as vacinas de RNAm do SARS-CoV-2, a proteção se inicia após
12 dias da primeira inoculação e se eleva em cerca de 50% de eficiência.
Após a segunda injeção três ou quatro semanas mais tarde, a segunda fase
da resposta imune começa, e as células B e T de memória aumentam e a
eficácia se eleva para cerca de 95%.
|
|
|
 Return to the
Virology section of Microbiology and Immunology On-line
Return to the
Virology section of Microbiology and Immunology On-line
 Voltar
à Seção Virologia do Microbiologia e Imunologia Online
Voltar
à Seção Virologia do Microbiologia e Imunologia Online
This page last changed on
Monday, February 08, 2021
Page maintained by
Richard Hunt
Please report any problems to
Richard Hunt
|


 Figura 2
Torovírus
Figura 2
Torovírus

 Figura 18
Figura 18




























