|
|
|
|
 |
 |
|
Dr. Gene Mayer |
BACTERIOLOGÍA |
INMUNOLOGÍA
|
MICOLOGÍA |
PARASITOLOGÍA |
VIROLOGÍA |
|
EN INGLÉS |
BACTERIOLOGÍA
– CAPÍTULO NUEVE
MECANISMOS DE REGULACIÓN GENÉTICA
Dr Gene Mayer
Traducción Dr. en C. Paula Figueroa
|
|
VA EL CAPÍTULO 10 |
|
VIDEOCONFERENCIA
EN INGLÉS
|
|
E-MAIL
DR PAULA FIGUEROA |
|
|
|
OBJETIVOS DE ENSEÑANZA
Discutir la estructura y la transcripción de los genes bacterianos.
Describir los mecanismos moleculares que las bacterias usan para regular
la actividad genética.
Comparar y contrastar los operones inducible y reprimible.
Describir los mecanismos moleculares involucrados en la represión
catabólica y la atenuación.
Discutir las maneras como las bacterias regulan la actividad enzimática. |
REGULACIÓN DE LA EXPRESIÓN GENÉTICA
Las
bacterias no producen todo el tiempo todas las proteínas que son capaces de
elaborar. En lugar de eso, ellas se adaptan a su medio ambiente y sintetizan
solo aquellos productos genéticos esenciales para sobrevivir en un medio
ambiente en particular.
Por
ejemplo, las bacterias no sintetizan las enzimas necesarias para la síntesis del
triptofano cuando hay una fuente abundante de triptofano en su medio ambiente.
Sin embargo, cuando el triptofano está ausente, las enzimas se sintetizan. De
manera similar, solo porque una bacteria lleva un gen de resistencia a un
antibiótico, no significa que ese gen se va a expresar. El gen de resistencia
solo se puede expresar cuando el antibiótico está presente en el medio ambiente
que rodea a la bacteria.
Normalmente las bacterias llevan a cabo un control de la expresión genética
regulando los niveles de la transcripción. En las bacterias, los genes con
función relacionada se encuentran generalmente adyascentes uno al otro y están
regulados en forma coordinada, (cuando uno se expresa se expresan todos). La
regulación coordinada de los genes agrupados se consigue mediante la producción
de un mRNA policistrónico (un mRNA largo conteniendo la información de muchos
genes). Por tanto, las bacterias son capaces de "sentir" su medio ambiente y
expresar el juego de genes necesario y más apropiado para el medio ambiente en
particular regulando la transcripción de ciertos genes. |
|
CONCEPTOS CLAVE
Coordinación de la expresión genética.
RNAm policistrónico
Promotor
Operón
Operón inducible
Inductor
Gene estructural
Gene regulador
Represor
Operador
Control negativo
Represión catabólica
proteína CAP
Control positivo
Operón reprimible
Co-represor
Apo-represor
Atenuación
Región Líder
Inhibición por retroalimentación (Feedback)
Modificación epigenética
 Figura 1 El operón de lactosa
Figura 1 El operón de lactosa
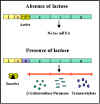 Figura 2 Transcripción de los genes lac en presencia y ausencia de
glucosa
Figura 2 Transcripción de los genes lac en presencia y ausencia de
glucosa |
Genes Inducibles - El Modelo del Operon
1.
Definición
Los genes inducibles son aquellos en los que la presencia de
una sustancia (un inductor) en el medio ambiente, enciende la expresión de
uno o más genes (genes estructurales) involucrados en el metabolismo de tal
sustancia ejemplos: la lactosa induce la expresión de los genes del
operon lac. Un antibiótico induce la expresión de un gen de resistencia.
La
inducción es común en las rutas metabólicas que dan como resultado el
catabolismo de una sustancia y el inductor es normalmente el sustrato de esa
ruta.
2. Operón de Lactosa
El operón de lactosa se ilustra en la Figura 1.
a.
Genes estructurales - El operón de lactosa contiene tres genes
estructurales que codifican para las enzimas involucradas en el metabolismo
de la lactosa. El gen lac z codifica para la β-galactosidasa, una
enzima que convierte la lactose en glucosa y galactosa, el gen lac y
que codifica para una permeasa, la cual está involucrada en la incorporación
de la lactosa, y el gen lac a, que codifica para una galactosa
transacetilasa. Estos genes se transcriben desde un promotor común en un
mRNA polícistrónico, el cual es traducido para dar lugar a las tres enzimas.
b.
Gen Regulador- La expresión de los genes estructurales no
solamente se ve influenciada por la presencia o ausencia del inductor,
también está controlada por un gen regulador específico. El gen regulador
puede estar cerca o lejos de los genes que están siendo regulados. El gen
regulador codifica para una proteína específica, un producto denominado
REPRESOR.
c.
Operador - El represor actúa mediante la unión con una región
específica del DNA llamada el operador, la cual está adyacente a los genes
estructurales que se están regulando. Los genes estructurales junto con la
región operadora y el promotor se conocen como un OPERÓN. Sin embargo, la
unión del represor con el operador se previene por el inductor y el inductor
puede también remover el represor que ya se haya unido al operador. No
obstante, en presencia del inductor el represor se inactiva y no se une al
operador, dando como resultado la transcripción de los genes estructurales.
En contraste, en ausencia del inductor, el represor si está activo y se une
al operador, lo cual da como resultado la inhibición de la transcripción de
los genes estructurales. Este tipo de control se conoce como CONTROL
NEGATIVO, ya que la función del producto del gen regulador (el represor) es
apagar la transcripción de los genes estructurales.
d.
Inductor – La transcripción de los genes lac está influenciada
por la presencia o ausencia de un inductor (lactosa u otros β-galactósidos)
(Figura 2).
|
Ej. |
+ inductor |
Expresión |
|
|
- inductor |
No hay expresión |
|
|
CHIME
 Haga click en la imagen a la izquierda para ver una estructura molecular
rotatoria del represor lac unido al DNA del operón lac.
Requiere Netscape y la conexión a Chime. Baje Chime
aquí)
Haga click en la imagen a la izquierda para ver una estructura molecular
rotatoria del represor lac unido al DNA del operón lac.
Requiere Netscape y la conexión a Chime. Baje Chime
aquí) |
 Figura 3 Represión
Catabólica
Figura 3 Represión
Catabólica
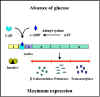 Figura 4 Efecto de la glucose en la expresión de las proteínas
codificadas por el operón lac
Figura 4 Efecto de la glucose en la expresión de las proteínas
codificadas por el operón lac
 Figura 5 Efecto de la glucosa en la expresión de las proteínas
codificadas por el operón lac
Figura 5 Efecto de la glucosa en la expresión de las proteínas
codificadas por el operón lac |
3. Represión Catabólica (Efecto de la Glucosa)
Muchos operones inducibles no solamente están controlados por sus
respectivos inductores y genes reguladores, sino que también se ven
controlados por el nivel de glucosa en el medio ambiente. La capacidad
de la glucosa en el control de la expresión de un número de diferentes
operones inducibles se le llama REPRESIÓN CATABÓLICA.
La cual se ilustra
en la Figura 3.
La represión catabólica se observa generalmente en aquellos operones que
están involucrados en la degradación de los componentes que se emplean
como fuentes de energía. Dado que la glucosa es la fuente preferida en
las bacterias, la capacidad de la glucosa para regular la expresión de
otros operones, asegura que las bacterias utilizarán glucosa antes que
cualquier otra fuente de carbono como fuente de energía.
Mecanismo
En las bacterias existe una relación inversa entre los
niveles de glucosa y los de AMP cíclico (AMPc). Cuando los niveles de
glucosa son bajos, los niveles de AMPc son altos y viceversa. Esta
interrelación existe porque el transporte de glucosa al interior de la
célula inhibe a la enzima adenilato ciclasa que es la que produce el
AMPc. En las bacterias el AMPc se une a una proteína de unión con AMPc
llamada CAP o CRP. El complejo AMPc-CAP y no la proteína CAP por sí
sola, es capaz de unirse a un sitio en los promotores de los operones
sensibles a la represión catabólica. La unión con el complejo hace al
promotor más eficiente, por lo tanto se llevarán a cabo más procesos de
iniciación de la transcripción de ese promotor, como se ilustra en las
Figuras 4 y 5. Debido a que el papel del complejo CAP-AMPc es encender la transcripción, este tipo de control se considera como
CONTROL POSITIVO. Las consecuencias de este tipo de control son que,
para que se logre la expresión máxima de un operón sensible a represión
catabólica, la glucosa necesariamente debe estar ausente en el medio
ambiente bacteriano y el inductor del operón deberá estar presente. Si
ambos están presentes el operón no se expresará a su nivel máximo hasta
que la glucosa sea metabolizada completamente. Obviamente, no hay
expresión del operón, es decir esta no ocurre a menos que el inductor se
encuentre presente.
|
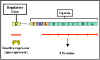 Figura 6 El operón de
triptofano
Figura 6 El operón de
triptofano
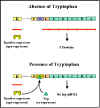 Figura 7 El efecto del triptofano sobre la expresión del operón trp
Figura 7 El efecto del triptofano sobre la expresión del operón trp
|
Genes Reprimibles - El Modelo del Operón.
1.
Definición
Los genes reprimibles son aquellos en los cuales la presencia
de una sustancia (un co-represor) en el medio ambiente, apaga la
expresión de uno o más genes (genes estructurales) involucrados en el
metabolismo de una sustancia.
Ejemplo, el triptofano reprime la expresión de los genes
trp.
La
represión es común en las rutas metabólicas que dan como resultado la
biosíntesis de una substancia y el co-represor normalmente es el producto
final de la ruta que está siendo regulada.
2.
Operón del triptofano
El operón para triptofano se ilustra en la Figura 6.
a.
Genes estructurales – El operón para el triptofano contiene
cinco genes estructurales que codifican para las enzimas involucradas en la
síntesis del triptofano. Estos genes se transcriben desde un promotor común
dando lugar a un RNA mensajero (mRNA) policistrónico, el cual se traduce
dando lugar a las cinco enzimas del operón.
b.
Gen Regulador- La expresión de los genes estructurales no
solamente se ve influenciada por la presencia o ausencia del co-represor, es
también controlada por un gen regulador específico. El gen regulador puede
estar cercano o lejano a los genes que están siendo regulados. Los genes
reguladores codifican para un producto protéico específico llamado el
REPRESOR (a veces también se le conoce como el apo-represor). Cuando el
represor se sintetiza es inactivo. Sin embargo, se puede activar al formar
un complejo con el co-represor (por ejemplo: el triptofano)
c.
Operador – El complejo represor/co-repressor actúa uniéndose a la
región específica del DNA llamada operador el cual es una secuencia
adyascente a los genes estructurales que se están regulando. Los genes
estructurales junto con la región del operador y el promotor se le conoce
como el OPERÓN. Por lo tanto, en presencia del co-represor, el represor se
activa y se une al operador, dando como resultado la represión de la
transcripción de los genes estructurales. En contraste, en ausencia del co-represor,
el represor es inactivo y por tanto no es capaz de unirse al operador, lo
cual da como resultado la transcripción de los genes estructurales. A este
tipo de control se le conoce como CONTROL NEGATIVO, ya que la función del
producto del gen regulador (represor) es apagar la transcripción de los
genes estructurales.
d.
Co-represor – La transcripción de los genes del triptofano está
influenciada por la presencia o ausencia de un co-represor (triptofano)
(Figura 7).
|
ej. |
+ co-represor |
no hay expresión |
|
|
- co-represor |
expresión |
|
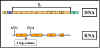 Figura 8 Mecanismo de atenuación
Figura 8 Mecanismo de atenuación
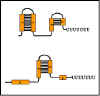 Figura 9 Formación de estructuras tallo-lupa
Figura 9 Formación de estructuras tallo-lupa |
3.
Atenuación
En
muchos operones reprimibles la transcripción que se inicia en el promotor puede
terminarse prematuramente en la region líder, que es la que precede al primer
gen estructural. (ej: la polimerasa termina la transcripción antes de
que se empiece a transcribir el primer gen del operón. Este fenómeno se conoce
como ATENUACION; la terminación prematura de la transcripción. Aunque la
atenuación se observa en un número de operones, el mecanismo se conoce y se
entiende mejor en aquellos operones que son reprimibles y que están involucrados
en la síntesis de los aminoácidos. En estos casos la atenuación se regula
mediante la disponibilidad del tRNA aminoacilado correspondiente al aminoácido
en cuestión.
Mecanismo (Ver la Figura 8 )
Aunque
la transcripción se inicia al nivel del promotor, realmente esta empieza antes del inicio del marco de lectura del primer gen estructural y en esta
región previa se forma un transcrito corto y se conoce como región líder. Esta
región líder contiene una señal de inicio y otra
de alto (paro) para la síntesis de proteínas. Ya que las bacterias no
tienen membrana nuclear, la transcripción y la transducción pueden ocurrir
simultáneamente. Por tanto se puede estar formando un péptido corto, al mismo
tiempo que la RNA polimerasa está transcribiendo la región líder. Este péptido de prueba contiene varios residuos de triptofano a la mitad del
mismo. Por lo tanto, si existe suficiente cantidad de triptofanil-t-RNA para
traducir el péptido de prueba, el péptido entero estará formado y el ribosoma
alcanzará la señal de alto. Por otra parte, si no hay suficiente
triptofanil-t-RNA para traducir el péptido, el ribosoma se arrestará en los dos
codones para el triptofano antes de alcanzar la señal de alto.
La
secuencia en el mRNA líder contiene cuatro regiones, las cuales tienen
secuencias complementarias entre sí (Figura 9). Por lo tanto, varias diferentes
estructuras de tallo y lupa se pueden formar. La región 1 solamente puede formar
pares de bases con la región 2; la region 2 puede formar pares de base ya sea
con la región 1 o con la región 3; la región 3 puede formar pares de bases con
las regiones 2 o 4; y la región 4 solamente puede formar pares de bases con la
región 3. Por lo tanto tres posibles estructuras de tallo/lupa pueden formarse
en el RNA.
región
1: región 2
región
2: región 3
región
3: región 4
Una de
las posibles estructuras (la región 3 que forma pares de bases con la región 4)
genera una señal para la RNA polimerasa para que ella termine la transcripción (ej.
para atenuar la transcripción). Sin embargo la formación de una estructura de
tallo y lupa puede ser que anule la formación de otras. Si la región 2 forma
pares de bases con la región 1 no estará disponible para parearse con la región
3. De manera similar, si la región 3 forma pares de bases con la región 2 no
estará disponible para formar pares de bases con la región 4.
La
capacidad de los ribosomas para traducir el péptido de prueba, afectará la
formación de las diferentes estructuras de tallo y lupa Figura 10. Si el
ribosoma alcanza la señal de alto de la traducción, ésta estará
cubriendo la región 2 y por tanto la región 2 no estará disponible para la
formación de pares de bases con otras regiones. Esto permite la generación de
la señal para la terminación de la transcripción, porque la región 3 estará
disponible para parearse con la región 4. Por lo tanto, cuando existe suficiente
triptofanil-t-RNA para traducir el péptido de prueba ocurrirá una atenuación y
los genes estructurales no se transcribirán. En contraste, cuando existe una
cantidad insuficiente de triptofanil-t-RNA para traducir el péptido de prueba,
entonces la atenuación sí ocurrirá. Esto es porque el ribosoma se detendrá en
los dos codones para triptofano de la región 1, permitiendo así que la región 2
forme pares de bases con la región 3 previniendo así la formación de la señal de
atenuación (ej. la región 3 se parearía con la región 4). Por lo tanto,
los genes estructurales se transcribirán.
|
 Figura 10 Mecanismo de atenuación
Figura 10 Mecanismo de atenuación |
| |
REGULACIÓN DE LA ACTIVIDAD ENZIMÁTICA.
Las
bacterias también tienen formas de regular las actividades de sus enzimas.
Inhibición por retroalimentación
Las actividades de las enzimas
bacterianas frecuentemente están sujetas a la regulación por retroalimentación
conocida como inhibición sujeta a retroalimentación (feedback). Normalmente, es
el producto final de la ruta metabólica el que actúa como inhibidor de la
primera enzima de la misma.
Es el paso en el cual se
lleva a cabo la regulación de la ruta metabólica.
Modificación epigenética
Las actividades de las enzimas bacterianas
también se regulan mediante modificaciones covalentes de las enzimas. Tales
modificaciones se llaman MODIFICACIONES EPIGENÉTICAS. Ejemplos. La adenilación de la sintetasa de glutamina, la
fosforilación
de la sintetasa de glicógeno.
Normalmente estas modificaciones son reversibles de manera que las actividades
de las enzimas pueden ser encendidas y apagadas.
|
|
|
 Regreso
a la Sección de Bacteriología de Microbiología e Immunología On-line Regreso
a la Sección de Bacteriología de Microbiología e Immunología On-line
El mantenimiento de ésta página es conducido por
Richard Hunt
|

 Figura 3 Represión
Catabólica
Figura 3 Represión
Catabólica
 Figura 6 El operón de
triptofano
Figura 6 El operón de
triptofano Figura 10 Mecanismo de atenuación
Figura 10 Mecanismo de atenuación





