|
|
 |
||||
|
Dr Richard Hunt |
BACTERIOLOGÍA | INMUNOLOGÍA | MICOLOGÍA | PARASITOLOGÍA | VIROLOGÍA |
|
|
|
||||

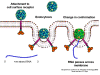
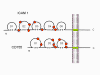
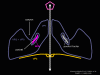
Figura 1a Estrategias de replicación de los virus de ARN
|
La mayoría de los virus de ARN tienen genomas de cadena sencilla, en vez de un ARN helicoidal de cadena doble (en contraste, los virus de ADN contienen ADN helicoidal doble, como el encontrado en el ADN de las células eucarióticas). Esto es porque el ADN es más fácilmente desenrollado durante la transcripción contrario al ARN de cadena doble que es muy estable (su temperatura de fusión es 10 grados más alta que la de un ADN de composición similar). Más aún, el desdoblamiento del ARN no es un mecanismo de la célula huésped, a diferencia del desdoblamiento del ADN que se lleva a cabo siempre que el ADN celular es transcrito a ADN o a ARNm.
Hay
dos tipos básicos de virus de ARN, aquellos que copian su ARN a un nuevo
ARN sin un ADN intermediario y aquellos que usan un ADN intermediario (figura
1a y b). El primer tipo incluye a los virus de ARN de sentido positivo
(i.e. picornavirus como el polio y rinovirus), virus de ARN de sentido
negativo (i.e. influenza) y virus de ARN de cadena doble (i.e.
rotavirus). Los virus de ARN que tienen un ADN intermediario son los
retrovirus (figura 1 a y c). Los virus animales de ARN codifican una polimerasa Puesto que el copiado de ARN a ARN no es una función de la célula huésped, los virus de ARN que no tienen una fase de ADN deben de codificar una ARN polimerasa ARN-dependiente (también llamada replicasa). Tales virus típicamente pasan por todo su ciclo vital en el citoplasma. Los retrovirus son virus de ARN de sentido positivo y deben codificar una ADN polimerasa ARN-dependiente (transcriptasa inversa) para sintetizar el provirus del ADN que luego es transcribido a ARN genómico por una enzima del huésped, la ARN polimerasa II. Dado que la ARN polimerasa II sintetiza ARNm en el núcleo de la célula huésped, el ciclo vital de los retrovirus tiene lugar mayormente en el núcleo. Hay una sola excepción al concepto de que todos los virus animales de ARN codifican su propia polimerasa y este es el agente de la hepatitis delta el cual no es un virus propiamente dicho. Este puede usar una enzima celular que, en su función normal copia ADN a ARN, para copiar su ARN a ARN.
|
||||
|
|
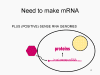
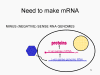
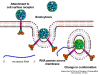
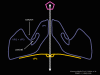
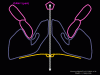
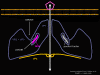
ARN mensajero viral Un virus de ARN necesita sintetizar ARN que funcione como un ARN mensajero para la síntesis proteica en una célula huésped. En el caso de los virus de ARN de sentido positivo (cuyo genoma, por definición, va en el mismo sentido que el ARNm), el ARN genómico puede servir como mensaje (figura 2). La cápside del virus funciona como el vehículo de transporte al citoplasma. Puesto que el genoma es ARN y es copiado por una ARN polimerasa, no hay necesidad de una secuencia promotora típica (caja TATA, caja CAT, etc.) corriente-arriba de los genes que codifican la proteína (como sucede con los virus de ADN o los retrovirus); incluso, los virus de sentido positivo no necesitan sintetizar nuevas proteínas antes de la síntesis de ARNm puesto que su ARN puede funcionar directamente como un mensaje. En contraste, las células infectadas por un virus de sentido negativo deben contener una proteína (la polimerasa) antes de que se pueda sintetizar ARNm de sentido positive y por tanto el virus debe de portar la polimerasa dentro de la cápside y llevarla a la célula infectada (figura 3). Además, el sentido de ARNm que es copiado del ARN genómico de sentido negativo entrante ha de tener las modificaciones normales del ARNm celular (chapa metilada, cola poliA) para que pueda ser traducido por la célula huésped. Esto significa que las enzimas modificadoras (metilada, GMP transferasa, polimerasa poliA) deben también ser empacadas en el virus entrante. Aun cuando la célula huésped pueda proveer estas enzimas, estarían localizadas en el núcleo (en donde el ARNm se sintetiza) y, como se ha hecho notar anteriormente, la mayoría de los virus de ARN sufren su ciclo vital en el citoplasma.
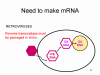
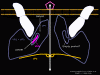
En
el caso de los retrovirus, el copiado de ARN a AND es necesario antes de
que pueda sintetizarse proteína, y por tanto la polimerasa (transcriptasa
inversa) debe también ser empacada en la particular viral (figura 4).
Estos virus de ARN, sin embargo, replican su genoma en el núcleo de la
célula huésped y por eso pueden usar las enzimas modificadoras de la
célula huésped. El problema del ARN monocistrónico
Las
células eucariotas tienen ARNm
monocistrónicos,
contrario a las bacterias que tienen ARNm
policistrónico.
Un ARNm monocistrónico codifica para una proteína puesto que el ribosoma
encuentra un punto de iniciación de traducción cerca del terminal 5’ del
ARNm y traduce la proteína hasta llegar a un codón de terminación. La
iniciación interna de la síntesis proteica dentro del ARNm no ocurre (excepto
en raras citaciones). Un ARN sencillo puede codificar para más de una
proteína en eucariotas pero esto se logra con empalmes de partes de la
transcripción original para crear otro ARNm que sirva de mensaje
monocistrónico. Las enzimas empalmadoras que llevan a cavo este proceso
se encuentran en el núcleo (dado que aquí es que se sintetiza en ARNm).
Puesto que los virus de ARN normalmente son citoplasmáticos, no pueden
aprovecharse de las enzimas empalmadotas. Por tanto, los virus de ARN
que solo contengan un ARNm deberían ser capaces de sintetizar solo una
proteína – pero han desarrollado un numero de trucos para superar esto (figura
5) y, de hecho, sintetizan mas de una proteína. Algunos se aprovechan de
las enzimas empalmadotas alternativas (y por tanto deben de tener una
fase nuclear). Otros sintetizan una única proteína que tiene actividad
proteasa; esta recorta en largo precursor en una serie de pequeñas
proteínas. Otros, como los picornavirus, han encontrado maneras de hacer
que un único ARNm funcione de manera policistrónica aun estando en una
célula eucariota. La forma en que logan esto será discutida mas adelante.
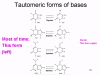
El problema de la corrección
Cuando se copia nuestro genoma de ADN, se hace una lectura de corrección
por la ADN polimerasa que corrige errores en la nueva copia de ADN. La
mayoría de los errores ocurre porque las bases normales que constituyen
el código en el ADN pueden existir en otras formas tautoméricas (figura
6). Estas extrañas formas pueden durar cortos periodos de tiempo pero si
en el momento en que se copia el ADN hay una base en su forma
tautomérica, esta será leída de forma incorrecta durante la síntesis de
ADN. De esta manera, A normalmente se aparea con T pero la forma
tautomérica de T se aparea con G y será copiada de esta manera. La
polimerasa de ADN regularmente no hacen lectura de corrección, y estas
mutaciones no son corregidas y son perpetuadas in generaciones futuras.
La tasa de mutación de la ARN polimerasa II es de 1 en 10,000 y por
tanto, para mantener a un bajo nivel la carga de mutación de los virus
de ARN, estos deben tener genomas pequeños; usualmente, los virus de ARN
tienen genomas de 10,000 nucleótidos o menos (en comparación, por
ejemplo, con los herpesvirus que tienen genomas de cerca de 150,000
nucleótidos). ¿Cómo es que todas estas mutaciones no vuelven inestables a los virus de ARN?
La
respuesta es que, en muchos casos, sí lo hacen. De esta manera, el VIH (que
tiene un genoma de poco menos de 10,000 nucleótidos) acumula muchas
mutaciones que resultan en polimorfismo poblacional y permiten al virus
convertirse resistente a los fármacos anti-virales. Sin embargo, muchos
virus de ARN (i.e. polio virus) son muy estables durante miles de años a
pesar de su alta tasa de mutación. La mejor manera de explicar esto es
con el concepto de idoneidad de forma física en un panorama Mulleriano.
De acuerdo a este concepto, ciertas cepas víricas pueden ser bien
adaptadas para replicarse en ciertos huéspedes (i.e. polio en humanos).
Pueden haber otras formas del virus que pudieran ser igualmente o más
exitosas pero para pautar a otra forma el virus tendría que pasar a
través de etapas menos adaptadas. De esta manera, cual el virus bien
adaptado es mutado, el virus mutante no puede competir y es eliminado.
En otras palabras, los virus bien adaptados tienen un pico de idoneidad
de su forma física en su nicho ecológico y son incapaces de cruzar el
valle hacia una adaptación más pobre (figura 7). Esto es algo similar a
las ardillas en los bordes norte y sur del Gran Cañón que se han
divergido puesto que no pueden adaptarse a las duras condiciones del
suelo del canon y no pueden, por tanto, ínter procrear. Consecuencias de un genoma de tamaño pequeño
Un
genoma pequeño restringe el número de proteínas que pueden ser
codificadas (figura 8). Se necesita la presencia de una proteína de
cápside para proteger el ARN y una polimerasa, que, como hemos visto,
debe de ser sintetizada por el virus. También será necesaria una
proteína de fijación para que el virus pueda infectar nuevas células. De
esta manera, habrá un número limitado de proteínas individuales aunque
los virus de ARN pueden ser bien largos por el uso de múltiples copias
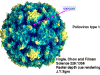
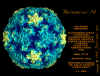
de diversas proteínas. Ciclo de vida Los Polio (figura 9) y rinovirus son picornavirus. Son virus de ARN pequeños, de sentido positivo, icosaédricos que no tienen envoltura (membrana bilipídica). Los picornavirus también incluyen los virus de la hepatitis A y enterovirus. El ciclo vital completo de estos virus ocurre en el citoplasma de la célula huésped. Puesto que el ARN tiene el mismo sentido que el ARNm, el ARN por sí mismo es infeccioso, aunque en menor medida que la partícula vírica completa. Esto es porque el virus tiene proteínas que se fijan a la superficie celular. Los Picornavirus se fijan a la célula huésped vía receptores específicos, al igual que casi todos los otros virus y son internalizados por la célula (figura 10). En el caso de los polio y rinovirus, esta es una proteína de función desconocida (en las células normales) denominada CD155, el receptor del polio virus o RPV. Si se tienen algunas nociones de la función normal de la CD155 de que se une a una proteína llamada vitronectina y por eso puede ser una proteína de adhesión implicada en la unión de una célula a su matriz extracelular. La mayoría de los rinovirus también pueden unirse a otro receptor que es una proteína de adhesión llamada CD54 o ICAM-1 (molécula de adhesión intercelular-1); el uso de estos receptores restringe el número de células que estos virus pueden infectar, delineando así su tropismo característico; por ejemplo, CD155 se expresa en muchos tipos de células incluyendo en el intestino y también las porciones terminales de los axones en las uniones neuromusculares, esto es en la membrana presináptica. CD155 e ICAM-1 son glicoproteínas que están ancladas a la membrana que atraviesan una sola vez (figura 11D). Sus regiones extracelulares, a las que se unen los picornavirus, tiene tres y cinco dominios tipo-inmunoglobulinas, respectivamente, con plicaturas tipo-inmunoglobulinas. En todo caso, el virus se une al dominio N-terminal. La ICAM-1 es más larga y protruye profundamente en el pliegue (figura 11C, D) mientras que la CD155 es más corta (figura 11E). Si se observa la estructura de la nucleocápside se verán doce pentones, uno en cada vértice del virus. Cada pentón está en contacto con otras cinco proteínas que lo rodean. Cada cara del virus tiene un complejo proteico que se asemeja a un trisquel o trinacria celta (estructura de tres piernas). Esto esta en contacto con otras seis proteínas (tres pentones y tres trinacrias y a veces se le llama hexón, aunque no parece un hexón sino un pentón. Puesto que el icosaedro tiene 20 caras, hay 20 trinacrias. Entre los pentones y las trinacrias hay una hendidura (figura 11A, B); el receptor en la superficie celular (ICAM-1 o CD155) se une en la hendidura (figura 11E) y precipita un cambio conformacional en algunas proteínas virales. La interacción de una célula y una proteína viral no es muy estable. En el caso de los rinovirus, la entrada al citoplasma parece seguir una endocitosis lo que sugiere que se requiere de un pH ácido para el cambio conformacional. Veamos con mejores detalles lo que sucede cuando un rinovirus se une a una célula mediante una ICAM-1. El ARN entrará a la célula a través de la membrana por el centro del pentón luego de que una proteína viral se incruste en la membrana plasmática. Si se hace una sección transversal a través del pentón (figura 12) (que tiene una morfología de 5 dobleces cuando se observa desde arriba), se puede observar que las moléculas VP1 forman la parte central del pentón y rodean un poro cerrado. La hendidura se localiza allí en donde la VP1 de adosa a VP2/3. Pero el piso de la hendidura no es plano. Hay un estrecho hoyo en el piso bajo el cual subyace un espacio mayor, conocido como el bolsillo. Normalmente, el bolsillo contiene “factor de bolsillo”. Este nunca ha sido aislado ni caracterizado pero aparenta ser una molécula larga de ácidos grasos en los estudios de cristalografía estructural. Debajo de las proteínas externas, VP1, VP2 y VP3, hay otra proteína vírica que no puede ser vista desde fuera (como en la figura 12) y esta es VP4. La Figura 13 muestra el esqueleto de las proteínas del pentón en una estructura más generalizada. La Figura 14 muestra la forma en la que se cree que la ICAM-1 encaja en el complejo VP1/2/3. Note que encaja en la hendidura pero no penetra el bolsillo. Se observa esto nueva vez en la figura 15 con el esqueleto proteico marcado. Una de las clases de fármacos que han sido usados para tratar las infecciones por picornavirus son las drogas WIN. Estas detienen el cambio conformacional que ocurre cuando el virus interactúa con la ICAM-1 y la figura 16 muestra lo que sucede. Las drogas WIN desplazan el “factor de bolsillo” y se acomodan arrimándose en el bolsillo (figura 17) y queda casi completamente enterrado como se pude ver en la imagen rellena del pentón (figura 18). Como se menciono anteriormente, una proteína, la VP4, está debajo de la proteína externa VP1/2/3. La VP4 tiene un ácido graso (ácido mirístico) unido a ella (figura 19) lo cual hace muy hidrofóbica a un terminal. En el modelo relleno (figura 20), se observa que el ácido graso protruye al poro formando un tapón en el medio del pentón. La VP3 también forma parte del tapón (figura 20). Cuando la ICAM-1 se fija a la hendidura, ocurre un cambio conformacional y la VP1 se inclina alejándose del centro del (figura 21). Esto exprime el bolsillo y es posible que esta unión desplace el factor de bolsillo. Ahora sabemos como funcionan las droga WIN. Se fijan al bolsillo con más afinidad que el factor de bolsillo y no son desplazadas cuando se une la ICAM-1. Por ende, el doblez para la apertura del poro no ocurre (figura 21b). Cuando la VP1 se inclina alejándose (figura 21b, c), el poro se abre puesto que la VP3 se desplaza de en medio. Esto permite a la VP4 moverse a través del poro (canal). El terminal amino de la VP4 tiene ácido mirístico (ácido graso) y esto de alguna manera se desplaza por el poro. Recuerde que el poro tiene un simetría de cinco dobleces y que realmente hay cinco de estas proteínas VP4 que son forzadas a través del canal. El ácido mirístico se incrusta en la bicapa de la membrana celular para formar un poro. La formación del poro se ayuda por el hecho de que la porción aminoterminal de la VP1 es una alfa hélice antipática que se libera de la unión a la ICAM-1. Como es de esperarse de la estructura tetramérica de VP1-4, la VP4 no debe de ser unida por anticuerpos anti-VP4 en el exterior del virus. Si se dejase el virus por mucho tiempo en solución, la VP4 seria tachada puesto que el virus “respira”. Cuando una de las drogas WIN se une al bolsillo dicha respiración se detiene demostrando que la unión más fuerte del fármaco al bolsillo estabiliza la estructura del virus. Como es de suponer, si se añade una parte de la ICAM-1 que se fije a la hendidura, habrá un cambio conformacional y el ARN sale del poro y el virus deja de ser infeccioso. El pentón no solo es la vía de salida del ARN a la célula que está siendo infectada, también es el sitio de entrada del ARN a la nucleocápside. |
||||
FILMACIÓN unión del rinovirus humano 14 a su receptor 
(832 Kb) Derechos reservados J.Y. Sgro (usado con autorización)
|
|||||

Figura 22 VpG está en el extreme 5' del genoma de ARN viral de sentido positivo pero se pierde antes de la traducción
|
TRADUCCIÓN Una vez que el ARN entra en la célula puede ser traducido. En el terminal 5’ del ARN hay una pequeña proteína viral llamada VPg (figura 22). Esta es removida del ARN cuando la célula es infectada (figura 22) y como el ARN tiene sentido positive, puede ser usado como ARNm. Regularmente, cuando un ARNm celular es traducido, la subunidad ribosómica menor reconoce la estructura chapada del terminal 5’ y luego el ribosoma ensambla después de la unión de la subunidad mayor. Sin embargo, no hay ninguna chapa metilada en el ARN de los picornavirus puesto que el chapamiento se da en el núcleo (por enzimas celulares) y el ciclo vital completo de los picornavirus sucede en el citoplasma. No obstante, la maquinaria celular de síntesis proteica si traduce el ARN viral. La Figure 23 muestra el complejo de iniciación normal que se forma en un ARNm chapado. En el ARN del picornavirus hay una región de estructura secundaria llamada sitio interno de entrada del ribosoma (IRES, por sus siglas en inglés) (figura 24). Este es reconocido por el complejo de proteínas que normalmente reconocen la chapa. Aparentemente, hay un factor celular adicional que es necesario para el reconocimiento del IRES pero no para chapa metilada normal de una ARNm. Es posible que la disponibilidad d estos factores del huésped determina en parte e tropismo de los picornavirus por ciertas células. Los Picornavirus también tienen actividad proteasa que puede separar una de las proteínas del complejo de iniciación, eIF4G (figura 25), y esto compromete seriamente la habilidad de la célula de traducir mensajes normales chapados pero afecta la traducción desde el IRES; por tanto, el virus puede suprimir la traducción celular mientras deja intacta la traducción de su propio ARN. El hecho de que solo haya un IRES significa que solo hay un producto primario de traducción; o sea, que solo puede sintetizarse una sola proteína. Esta proteína será eventualmente dividida en varias proteínas en el virus maduro y por ello, a esta proteína primaria se le llama policistrónica puesto que codifica más de un gen (cistrón). Las proteasas que dividen la poliproteína original son codificadas por el genoma viral y el proceso proteolítico se muestra en la figura 26.
|
||||
 Figura 24 En el ARN del picornavirus hay una región de estructura secundaria llamada sitio interno de entrada de ribosoma (IRES, por sus siglas en inglés)
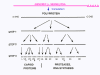 Figura 26 Las proteasas que separan la poliproteína original son codificadas en el genoma viral y el proceso proteolítico es ordenado |
|||||
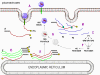 A
A
|
REPLICACIÓN DEL ARN Esto es muy simple si se le compara con otros virus de ARN. Ya que los picornavirus pasan todo su tiempo en el citoplasma, han de codificar una polimerasa (replicasa) sintetizada de la cadena de sentido del virus infectante. La polimerasa copia la cadena de sentido positivo a una de anti-sentido que luego es vuelta a copiar en cadena de sentido positivo que es empacada dentro del virus (figura 27). La replicación de ARN parece ocurrir en la superficie citoplasmática de las vesículas membranosas a las que se une la ARN polimerasa. Estas aparentemente salen del retículo endoplásmico, tal como las vesículas de transporte en las células no infectadas y las proteínas de membrana del cuerpo de Golgi. Sin embargo, cuando la célula es infectada por un picornavirus, las vesículas no se funden con la fase cis del cuerpo de Golgi como lo hacen las vesículas de transporte (figura 28). Estas vesículas tienen proteínas dianas específicas en sus superficies citoplásmicas (llamadas proteínas COP) y es posible que se involucren en la respuesta a la infección viral. Debe de recordarse que los picornavirus no tienen una envoltura lipídica y no tienen una glicoproteína de superficie. Por tanto, la producción del virus no es inhibida con el compromiso de la función del cuerpo de Golgi, como seria el caso con un virus envuelto. Se desconoce la razón de esta asociación a membrana de la replicación del ARN de los picornavirus pero se pueden concentrar varios sustratos en la vecindad de la polimerasa (recuérdese que en las bacterias, la replicación del ADN está asociada a membrana). El ARN de los picornavirus es poliadenilado en el terminal 3’, al igual que los ARNs mensajeros pero esta poliadenilación ocurre de diversas maneras. En la síntesis de ARNm de la célula huésped, la secuencia poliA no está codificada en la copia de ADN de este gen sino que es añadida por una enzima llamada polimerasa poliA usando ATP como sustrato. En el caso del ARN de los picornavirus, no obstante, la secuencia de poliA en la cadena de sentido positivo es copiada a una secuencia poliU en el terminal 5’ de la cadena de sentido negativo. Esto es copiado de nuevo a poliA en terminal 3’, también por la replicasa.
|
||||
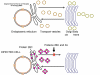
Figura 28 Cuando la célula es infectada por un picornavirus, las vesículas de transporte del retículo endoplásmico al cuerpo de Golgi no se funden con la fase cis del cuerpo de Golgi |
ENSAMBLAJE Como se mencionó anteriormente, las proteínas de los picornavirus son policistrónicas porque solo hay un sitio interno de unión de ribosoma. Esto significa que el virus puede ser ensamblado al tiempo que las diversas proteínas son clivadas de la poliproteína. La poliproteína primero es dividida en tres proteínas (P1, P2, P3). P1 es luego clivada en tres proteínas (VP0, VP1, VP3) que son las subunidades de la envoltura viral. Esto se lleva a cabo por proteasas virus-específicas que son parte de la poliproteína y son catalíticas como parte del producto primario de traducción. VP0, VP1 y VP3 se ensamblan en la subunidad estructural 5S (protómero). VP0 solo se deriva en VP2 y VP4 cuando el virus es ensamblado. Cinco de estos protómeros ensamblan en un pentámero 14S y doce pentámeros forman la procápside. El ARN es encapsulado en la procápside para formar un provirión. En esta etapa la VP0 es derivada en VP2 y VP4 y el virión está maduro, una particular vírica infecciosa. La VP0 solo es derivada sien ARN ha sido encapsidado. Las otras dos partes del producto primario de traducción (P2 y P3) son clivadas para formar un número de proteínas no estructurales (i.e. proteína que no se encuentran en la particular vírica madura pero que se usan durante la replicación en la célula infectada). Estas incluyen la replicasa y proteínas que alteran el metabolismo de la célula huésped. VPg, la proteína que se encuentra en la terminal de las moléculas genómicas de ARN de sentido positive, se forma a partir de P3. |
||||
|
LIBERACIÓN Muchas partículas de picornavirus se acumulan en el citoplasma y la célula muere resultando en lisis celular. |
|||||
|
De esta manera todo el ciclo vital del polio virus ocurre en el citoplasma
|
|||||
|
|||||